Tóm tắt
Vi bào tử trùng Enterocytozoon hepatopenaei (EHP) là một mầm bệnh mới, gây ra thiệt hại nặng nề về kinh tế trong ngành tôm He (Penaeus spp.) trên toàn thế giới. Để ngăn ngừa bệnh trên tôm, người ta đã đề xuất sử dụng hệ vi sinh vật đường ruột. Tuy nhiên, cần phải có kiến thức về hệ vi sinh vật và vật chủ. Chúng tôi đã đánh giá sự thay đổi của hệ vi sinh vật (vi khuẩn / nấm) và các chức năng của nó trong quá trình nhiễm EHP ở tôm được thử nghiệm trong 30 ngày bằng cách sử dụng giải trình tự rRNA 16S thông lượng cao và giải trình tự amplicon ITS. Mức độ nhiễm trùng được đánh giá lần đầu tiên bằng mô bệnh học kỹ thuật số định lượng. Dựa theo mức độ lây nhiễm, các nhà nghiên cứu đã phân chia bệnh thành 3 giai đoạn: giai đoạn đầu, giai đoạn phát triển và giai đoạn cuối. Trong giai đoạn đầu, EHP dường như không được phát hiện, và người ta đã tìm thấy sự đa dạng cao của các vi sinh vật có lợi liên quan đến quá trình đồng hóa chất dinh dưỡng. Trong giai đoạn phát triển của bệnh, hầu hết tôm bắt đầu có mức độ nhiễm bệnh cao liên quan đến sự suy giảm các vi sinh vật có lợi và sự gia tăng các loại nấm cơ hội / gây bệnh. Trong giai đoạn cuối, tôm có các biểu hiện về mức độ nhiễm bệnh khác nhau, cho thấy vị trí của các vi sinh vật có lợi đã bị chiếm chỗ bởi vi khuẩn cơ hội / gây bệnh và nấm liên quan đến quá trình lây nhiễm mầm bệnh và cạn kiệt nguồn năng lượng dự trữ. Mô hình suy giảm theo chu kỳ của EHP và ảnh hưởng của nó đối với các vi sinh vật có lợi và các chức năng có lợi của hệ vi sinh vật gan tụy tôm đã được thảo luận.
Giới thiệu
Tôm thẻ chân trắng (Penaeus vannamei) là loài tôm nuôi quan trọng về kinh tế, chiếm 52,9% tổng sản lượng nuôi giáp xác (9,4 triệu tấn) trên toàn thế giới. Mặc dù sản lượng tăng đều trong những năm gần đây, nhưng nghề nuôi tôm vẫn luôn phải đối mặt với các thách thức do sự xuất hiện và lây lan của các mầm bệnh mới như bệnh nhiễm vi bào tử trùng (HPM), gây ra bởi vi bào tử trùng Enterocytozoon hepatopenaei (EHP). Bệnh này đã được báo cáo lần đầu tiên vào năm 2001, ảnh hưởng đến tôm sú P. monodon và được gọi là hội chứng tăng trưởng chậm (MSGS). Năm 2009, EHP chính thức được mô tả từ tôm post / ấu trùng sú P. monodon và bệnh này sau đó được gọi là HPM.
Bệnh nhiễm vi bào tử trùng hiện đã được báo cáo ở Thái Lan, Việt Nam, Ấn Độ, Trung Quốc, Indonesia, Malaysia, Venezuela và tiếp tục lây lan sang các nước nuôi tôm khác. Hiện nay, EHP được coi là một trong những mối đe dọa lớn đối với nghề nuôi tôm và đang gây ra những hạn chế lớn đối với ngành tôm trên khắp châu Á. Vì các biện pháp an toàn sinh học trước đây không thể ngăn chặn sự lây lan của mầm bệnh trong nuôi tôm và chỉ có một số phương pháp có sẵn để điều trị các bệnh ở giáp xác, do đó, các nhà nghiên cứu đã tập trung nỗ lực vào việc khám phá các giải pháp thay thế mới để ngăn ngừa bệnh ở tôm, bao gồm cả việc điều chỉnh hệ vi sinh vật trên vật chủ. Cuối cùng, một giải pháp thay thế đầy hứa hẹn đã ra đời, vì hệ vi sinh vật có liên quan chặt chẽ đến các chức năng cụ thể của vật chủ và có thể được sử dụng như một công cụ để thành công trong nuôi tôm, giúp ngăn ngừa / kiểm soát sự lây nhiễm EHP.
Việc điều chỉnh hệ vi sinh vật đã được sử dụng để cải thiện sức khỏe, năng suất tăng trưởng và khả năng kháng bệnh ở tôm, từ đó hỗ trợ nâng cao sản xuất trang trại. Ở cấp trang trại, việc điều chỉnh này có thể được thực hiện bằng cách sử dụng probiotics, prebiotics, synbiotics hoặc tạo ra môi trường thuận lợi cho probiotics (ví dụ: biofloc). Tuy nhiên, để điều chỉnh hệ vi sinh vật hiệu quả và thúc đẩy tăng trưởng đòi hỏi phải có kiến thức về sức khỏe và hệ vi sinh vật ở tôm bệnh.
Hệ vi sinh vật của một sinh vật có thể được giải mã bằng cách sử dụng công nghệ giải trình tự thế hệ mới thông qua việc khuếch đại và giải trình tự các vùng siêu biến của gen như rRNA 16S cho vi khuẩn và trình tự ITS cho nấm. Giải trình tự amplicon bằng các phương pháp giải trình tự thế hệ mới cho phép định lượng mức độ phong phú, đánh giá sự đa dạng và dự đoán các chức năng của hệ vi sinh vật dựa trên đơn vị phân loại chúng. Các phương pháp này đã được nhiều nhà nghiên cứu sử dụng để thu được bản đồ có độ phân giải cao đối với hệ vi sinh vật chủ. Hơn nữa, các phương pháp này cho phép hiểu rõ hơn về sự tương tác phức tạp giữa vật chủ và vi sinh vật khi tình trạng sức khỏe của vật chủ bị ảnh hưởng bởi sự hiện diện của mầm bệnh.
Vẫn còn rất ít dữ liệu đề cấp đến việc điều chỉnh hệ vi sinh vật gan tụy (vi khuẩn và nấm) cũng như các chức năng liên quan của EHP ở tôm thẻ P. vannamei. Nghiên cứu này đánh giá thành phần, sự đa dạng và chức năng của hệ vi sinh vật gan tụy ở tôm thẻ chân trắng P. vannamei bị nhiễm EHP trong 30 ngày sau khi cảm nhiễm với mầm bệnh. Các phát hiện đã cung cấp những dữ liệu có giá trị về các mô hình suy giảm theo từng giai đoạn bệnh trong hệ vi sinh vật gan tụy tôm khi nhiễm EHP.
Phương pháp
Thu thập tôm
Tôm thẻ chân trắng P. vannamei không có mầm bệnh cụ thể (SPF), trọng lượng trung bình ~ 5 g có nguồn gốc từ Viện Đại dương (Oahu, Hawaii, Hoa Kỳ) đã được sử dụng cho thử nghiệm sinh học, thực hiện tại Phòng thí nghiệm Bệnh học Nuôi trồng Thủy sản của Đại học Arizona. Tất cả tôm trong nghiên cứu đều được chứng nhận SPF và liên tục được kiểm tra âm tính với các mầm bệnh cả được liệt kê và không liệt kê trong OIE, bao gồm cả EHP. Trước khi thử nghiệm, tôm được thuần trong hệ thống nuôi tuần hoàn 100L (25°C) trong 2 tuần, sục khí nhẹ và cho ăn với chế độ ăn thương mại (Rangen, 40% protein).
Thử nghiệm cảm nhiễm
Thử nghiệm cảm nhiễm được thực hiện theo phương pháp “sống chung”. Tức là, khi thuần, tôm SPF được phân biệt bằng cách cắt một phần nhỏ của đuôi. Trước khi tôm SPF tiếp xúc với tôm nhiễm EHP (ngày 0), có tổng cộng 2 con tôm SPF được lấy mẫu và mô gan tụy được mổ vô trùng, một phần được đông lạnh nhanh trong nitơ lỏng và phần còn lại được cố định trong cồn Davison dung dịch -formalin-axit axetic (AFA). Ngày hôm sau, có tổng cộng 36 con tôm SPF được chuyển sang bể thí nghiệm chứa tôm nhiễm EHP. Thử nghiệm sống chung được thực hiện trong 30 ngày, trong khi những con tôm trong thí nghiệm được lấy mẫu trong suốt thời gian cảm nhiễm. Cảm nhiễm EHP bằng phương pháp cho sống chung với nhau là khá phổ biến, vì đây là phương pháp nhanh chóng và tiết kiệm chi phí để lây lan ký sinh trùng, mô phỏng cụ thể các con đường lây nhiễm EHP tự nhiên trong trang trại nuôi tôm.
Trong quá trình thử nghiệm, tôm được cho ăn thức ăn viên thương mại hàng ngày (Rangen, 40% protein). Nhiệt độ và độ mặn trong bể lần lượt ở mức 25°C và 33 ppu. Trong toàn bộ thời gian của thử nghiệm thách thức, vào mỗi ngày thứ ba, 2-3 tôm có ký hiệu SPF được lấy mẫu. Mô gan tụy được cắt thành hai phần bằng nhau, một phần được đông lạnh nhanh và giữ ở nhiệt độ – 80°C để phân tích hệ vi sinh vật, phần còn lại được cố định trong dung dịch Davidson AFA để phân tích lai tại chỗ. Mô gan tụy được chọn làm mô đích vì EHP phát triển và nhân rộng trong mô này. Nghiên cứu này được thực hiện theo các quy trình tiêu chuẩn của Phòng thí nghiệm Bệnh học Nuôi trồng Thủy sản của Đại học Arizona, phòng thí nghiệm này đã được USDA và ISO (17043 và 170225) công nhận về Chẩn đoán Bệnh ở Giáp xác.
Mô bệnh học định lượng sử dụng lai tại chỗ
Mục đích của phương pháp này là đánh giá mức độ lây nhiễm của EHP đối với tôm SPF sau thử nghiệm sống chung. Lai tại chỗ (ISH) được thực hiện theo các phương pháp đã được báo cáo trước đây để phát hiện EHP. Trong đó, các đoạn mồi đặc hiệu EHP 510 F (5′-GCCTGA GAG ATG GCT CCC ACG T-3′) và 510 R (5′-GCG TAC TAT CCC CAG AGC CCG A-3′) được sử dụng và nối đuôi với digoxigenin-11-dUTP (Integrated DNA Technologies®, San Diego, CA, USA) ở cuối đoạn 3′. Mức độ nghiêm trọng của nhiễm EHP được phân loại từ G0-G4 theo sơ đồ phân loại bán định lượng.
Tóm lại:
Cấp 0: không có dấu hiệu nhiễm trùng.
Cấp 1: có dấu hiệu nhiễm bệnh nhưng ở mức độ có thể thấp hơn so với mức lâm sàng.
Cấp 2: mức độ nhiễm trung bình, thể hiện bằng số lượng và mức độ nghiêm trọng của mầm bệnh.
Cấp 3: mức độ nhiễm từ trung bình đến cao, thể hiện qua số lượng và mức độ nghiêm trọng của mầm bệnh.
Cấp 4: số lượng lớn mầm bệnh gây ra tổn thương và phá hủy mô.
Các lam kính ISH có chứa vùng mô bị bóc tách được quét và kỹ thuật số hóa bằng Máy quét MoticEasyScan Pro 6 Pathology Slide Scanner (Meyer Instruments, Inc., TX, USA). Hình ảnh kỹ thuật số được xử lý bằng Image-Pro Plus 10.0 (Media Cybernetics, Bethesda, MD, USA) theo López-Carvallo và cộng sự. Trong mỗi trang chiếu, 3 hình ảnh ngẫu nhiên ở độ phóng đại 10 × được chọn đại diện cho sự lây nhiễm của một con tôm cụ thể. Thông tin này được sử dụng để tính toán chỉ số nhiễm EHP (EII) bằng công thức sau:
EII (%) = ((CAE / TCA) × 100)
trong đó: CAE là vùng bao phủ của EHP (µm2)
TCA là tổng vùng bao phủ của vùng được phân tích của mô bị ảnh hưởng (µm2).
Các tỷ lệ phần trăm EII chỉ định cho mức độ nghiêm trọng của mầm bệnh được đề xuất bởi Lightner. Tỷ lệ phần trăm EII được bình thường hóa bằng arcsin và phân tích phương sai một chiều được thực hiện để đánh giá sự khác biệt đáng kể của mức độ nhiễm trùng và giai đoạn của bệnh.
Phân lập DNA của vi khuẩn và nấm
Tổng cộng 30 mẫu gan tụy đông lạnh nhanh từ tôm SPF được đánh dấu, nuôi chung với tôm nhiễm EHP đã được sử dụng để tách chiết DNA bộ gen của vi khuẩn và nấm (gDNA) từ mỗi con. Tổng gDNA được tách chiết từ mô được lấy mẫu (20 mg) bằng Bộ Kit tách chiết Genomic DNA (Norgen Biotek Corp®, ON, Canada) và Bộ tinh lọc DNA Maxwell® 16 Cell LEV (Promega, WI, Hoa Kỳ) theo nhà sản xuất hướng dẫn.
Tính toàn vẹn của tổng gDNA được chứng thực bằng điện di trên gel, chất lượng và nồng độ được đo bằng NanoDrop 2000 / 2000c ™ (Thermo Fisher Scientific, Waltham, MA, USA). DNA phân lập được gửi cho NGS tại OmegaBioservices, Norcross, GA. Phản ứng PCR được thực hiện bằng cách sử dụng mồi 16S rRNA IlluminaF (5′-CCTACGGGNGGCWGCAG-3′) và IlluminaR (5′-ACTACHVGGGTATC-TAATCC-3′) trong khi bộ đệm phiên mã bên trong (ITS) được khuếch đại bằng cách sử dụng cặp mồi ITS1F (5′-C TTG GTC ATT TAG AGG AAG TAA-3 ′) và ITS4R (5′-T CCT CCG CTT ATT GAT ATG C-3′).
Giải trình tự các amplicon 16S rRNA V3-V4 và ITS
Trước khi xây dựng các hệ thống dữ liệu, chất lượng gDNA đã được đánh giá bằng TapeStation (Agilent®). Có tổng 30 hệ thống dữ liệu được xây dựng để phân tích vi khuẩn và 30 hệ thống dữ liệu được xây dựng để phân tích nấm. Tất cả đã được chuẩn bị bằng cách sử dụng bộ kit KAPA HyperPlus (Roche®, Basile, Thụy Sĩ) theo khuyến nghị của nhà sản xuất. Chất lượng hệ thống dữ liệu được phân tích bằng hệ thống Agilent Bioanalyzer 2100 (Agilent®, Santa Clara, CA, USA) và được giải trình tự với nền tảng Illumina Mi-Seq® (San Diego, CA, USA) ở chế độ đầu cặp (2X300). Việc tạo ra các hệ thống dữ liệu và giải trình tự được thực hiện bởi OmegaBioservices (Norcross, GA, USA). Tổng cộng 1.331.020 16S rRNA V3 – V4 và 883.460 ITS đọc thô được tạo ra thông qua giải trình tự.
Bioinformatics (Tin sinh học) và phân tích thống kê
Để tăng số lần sử dụng các thông số khác nhau trong việc đánh giá hệ vi sinh vật của tôm P. vannamei nhiễm EHP, chúng tôi đã sử dụng công cụ giao diện đồ họa người dùng có tên EasyMAP17. Công cụ này tích hợp Thông tin chi tiết định lượng về Hệ sinh thái vi sinh vật 2 (QIIME2, phiên bản 2018.4.0), Kích thước hiệu ứng phân tích tuyến tính (LefSe, phiên bản 1.0.8.post1) và Điều tra phát sinh loài của các quần thể bằng đường ống Reconstruction of Unobserved States (PICRUSt, phiên bản 1.1 .3), và kết hợp cơ sở dữ liệu của Greengenes, SILVA và UNITE. Cơ sở dữ liệu được tạo ra thông qua giải trình tự thông lượng cao để phân tích vi khuẩn và nấm, chúng được phân tích riêng biệt bằng cách sử dụng cùng một đường ống ngoại trừ cơ sở dữ liệu. Cơ sở dữ liệu Greengenes được sử dụng để xác định đơn vị phân loại vi khuẩn và UNITE được sử dụng để xác định đơn vị phân loại nấm. Các bài đọc thô lần đầu tiên được phân tích bằng phần mềm FastQC26 để đánh giá chất lượng. Các hệ thống dữ liệu kết thúc cặp chứa tệp đọc thô, siêu dữ liệu mẫu và tệp kê khai đã được tải lên tại cổng EasyMAP (http://easymap.cgm.ntu.edu.tw/ ). Các tệp này được sử dụng để xác định các hệ thống dữ liệu và xác định nhóm thống kê cho mỗi mẫu để phân tích sau.
Đối với tất cả các phân tích thống kê, chúng tôi sử dụng ba nhóm theo phân loại giai đoạn bệnh (đầu, phát triển và cuối), trong đó các sinh vật riêng lẻ được coi là bản sao sinh học. Quyết định này được đưa ra dựa trên nhiều phân tích nhóm, vì người ta đã xác nhận rằng các sinh vật trong cùng một nhóm có liên quan đến các điều kiện sinh lý, mô hoặc môi trường tương tự nhau. Mối tương quan của Pearson và PCoA chủ yếu là duy trì sự phân nhóm theo giai đoạn bệnh so với mức độ nhiễm bệnh và thời gian tiếp xúc với mầm bệnh.
Vì các tệp trình tự đã được phân kênh, nên tệp kê khai được sử dụng theo nền tảng EasyMAP. Trình cắm QIIME2 q2-dada2 được sử dụng để cắt bớt phần đọc chất lượng thấp (Q <25), hợp nhất các phần đọc chất lượng cao theo cặp và loại bỏ các chuỗi ký tự số. Không có trình tự số nào được sử dụng cho đơn vị phân loại loài (OTU). Phân tích loài (xây dựng bảng OUT, phân loại loài, bảng số liệu tương đối theo đơn vị phân loại) được đánh giá bằng cách sử dụng trình phân loại q2-feature-classifier của trình cắm thêm QIIME2. Có đến 97% sự giống nhau về trình tự đã được xem xét để phân nhóm trình tự và các trình tự tương tự từ cùng một đơn vị phân loại được nhóm lại thành một OTU. Quá trình tạo cây phát sinh loài được thực hiện bằng cách cắm q2-alignment và q2-phy phát sinh. Biểu đồ thanh tần số cho phyla và bản đồ nhiệt đã được sử dụng để hình dung các chi được vẽ trong R.
Phân tích độ phong phú vi phân phân loại được thực hiện bởi trình cắm thêm LEfSe sử dụng cơ sở dữ liệu KEGG ở cấp độ ba và thử nghiệm Wilcoxon (α <0,05). Mức độ phong phú của phân loại phân loại chỉ được đăng ký khi LDA> 2. Mức độ phong phú của phân loại được biểu thị theo phân biệt được vẽ bằng cách sử dụng đồ họa thanh, bản đồ nhiệt và biểu đồ hình tròn được cung cấp bởi nền tảng EasyMAP hoặc được vẽ bằng R.
Nền tảng EasyMAP đã cung cấp kết quả thống kê về sự đa dạng alpha và beta. Các OTU được quan sát, chỉ số đa dạng Shannon (H) và chỉ số đa dạng Simpson (D) được sử dụng làm công cụ thống kê tiêu chuẩn về đa dạng alpha và Phân tích tọa độ chính (PCoA) để phân tích đa dạng beta. Sự đa dạng alpha được đánh giá bởi Kruskal – Wallis của tất cả các nhóm và theo cặp; (P <0,05) và được trực quan hóa bằng cách sử dụng các ô hộp. Sự đa dạng beta được đánh giá bởi PERMANOVA (P <0,05) sử dụng phương pháp Jaccard (đo lường sự hiện diện / không hiện diện) và PCoA được sử dụng để hình dung thành phần vi sinh vật và mức độ giống / khác nhau giữa các mẫu.
Chức năng microbiome được dự đoán bằng cách sử dụng sự đa dạng của các đơn vị phân loại được biểu thị khác biệt và trình cắm thêm PICRUSt ánh xạ ID Greengenes với đường dẫn KEGG tương ứng. Công cụ này đã được sử dụng để dự đoán thành phần chức năng của vi khuẩn trong ruột của động vật giáp xác. Chức năng dự đoán của hệ vi sinh vật được vẽ bằng đồ họa hình tròn sử dụng điểm LDA log10.
Bản đồ nhiệt, biểu đồ cột và tròn, và phân cụm phân cấp (tương quan Pearson) được thực hiện bằng cách sử dụng ggplot2 và hclus tương ứng, sử dụng phần mềm R trong Rstudio. Mức độ phong phú tương đối được thể hiện trong các hình bản đồ nhiệt đã được biến đổi log2 để hình dung các đơn vị phân loại có mức độ phong phú tương đối thấp (<1%). Thiết kế thử nghiệm chung có thể được hình dung như một bản tóm tắt đồ họa để giúp người đọc dễ hiểu (Hình 1). Đối với tất cả các bản sao, một ID mô tả đã được thêm vào bao gồm ngày lấy mẫu (D0 – D30), cấp độ nhiễm (I0 – I4) và số lượng bản sao (R1 – R3) để tạo điều kiện theo dõi những thay đổi của hệ vi sinh vật do nhiễm EHP (ví dụ: D0_I0_R1). Ngoài ra, các nhóm thống kê (giai đoạn bệnh) được đánh dấu bằng ba màu khác nhau (Đầu: xanh lá cây; Phát triển: đỏ; Cuối: xanh lam) để phân tích vi khuẩn và nấm. Đặc biệt ở nấm, phân tích dự đoán chức năng vi sinh và biểu hiện khác biệt được thực hiện bằng cách sử dụng các phân nhóm đã được phân chia trong nhóm đối chứng, giai đoạn đầu: D1N, D2N, D2I; giai đoạn phát triển: D3E D3L, D4E và D4L; giai đoạn cuối: D5E và D5L. Nhóm đối chứng tương ứng với những con tôm được lấy mẫu vào ngày thứ 3; D1 vào ngày 6; D2 vào ngày 9 và 12; D3 vào ngày 15 và 18; D4 vào ngày 21 và 24; D5 vào ngày 27 và 30. “N” được sử dụng để mô tả những con tôm không bị nhiễm bệnh, “I” đại diện cho những con bị nhiễm ở giai đoạn đầu, “E” đại diện cho những con có mức nhiễm I1 và I2, và “L” cho những có mức nhiễm I3 và I4. Các nhóm phụ này được đánh giá trong các đơn vị phân loại nấm do mô hình biến thiên giữa các mẫu.
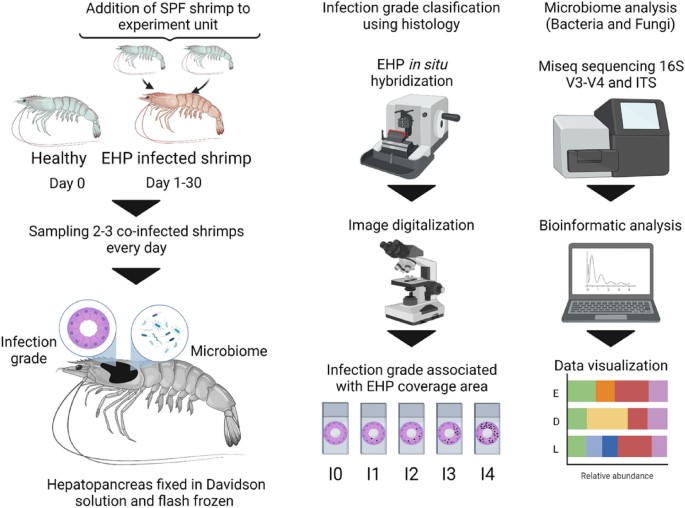
Hình 1: Sơ đồ nghiên cứu và thu thập mẫu. Thiết kế thí nghiệm để đánh giá cấu trúc quần thể vi khuẩn và nấm, cũng như dự đoán chức năng hệ vi sinh vật gan tụy ở tôm nhiễm EHP trong 30 ngày.
Kết quả
Mô bệnh học kỹ thuật số: phát hiện EHP và định lượng mức độ nghiêm trọng của bệnh
5 cấp độ nhiễm khuẩn gan tụy của tôm sau khi cảm nhiễm với mầm bệnh được phân tích bởi Lightner: I0, I1, I2, I3 và I4 có liên quan (P <0,05) với giá trị EII lần lượt là 0%, 0,004–0,14%, 0,51–5,92%, 6,85–9,26% và 10,15–45,67% (Hình 2). Có 3 giai đoạn bệnh được sử dụng để mô tả chu kỳ lây nhiễm: giai đoạn đầu, đang phát triển và giai đoạn cuối (Hình 3A) được hỗ trợ bởi EII và được đánh giá theo mức độ nhiễm bệnh (I0 – I4), theo từng giai đoạn bệnh (đầu-cuối) (Hình 3B), và giá trị trung bình cho từng giai đoạn bệnh (Hình 3C).
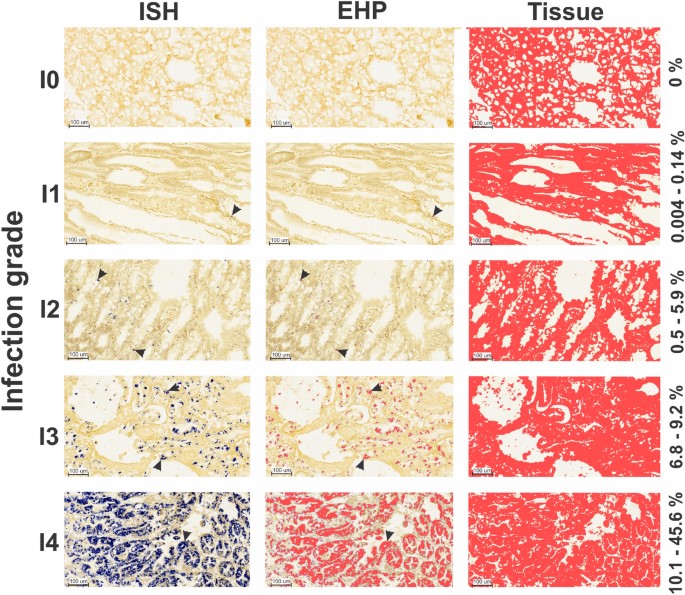
Hình 2: Thang đo cấp độ nhiễm EHP. Cấp độ nhiễm bệnh được đánh giá bằng phương pháp lai tại chỗ (ISH) sử dụng chỉ số nhiễm trùng EHP (EII) và được phân tích theo phương pháp do Lightner đề xuất. Mỗi cấp độ (I0 – I4) đại diện cho một dải giá trị EII (%) được hiển thị ở phía bên phải của ảnh. Hình ảnh cho thấy EHP và vùng mô được định lượng riêng biệt bằng cách sử dụng hình ảnh được phóng to lên gấp 10 lần để xem EII theo từng giai đoạn nhiễm bệnh. Mũi tên đen đánh dấu sự có mặt của EHP.
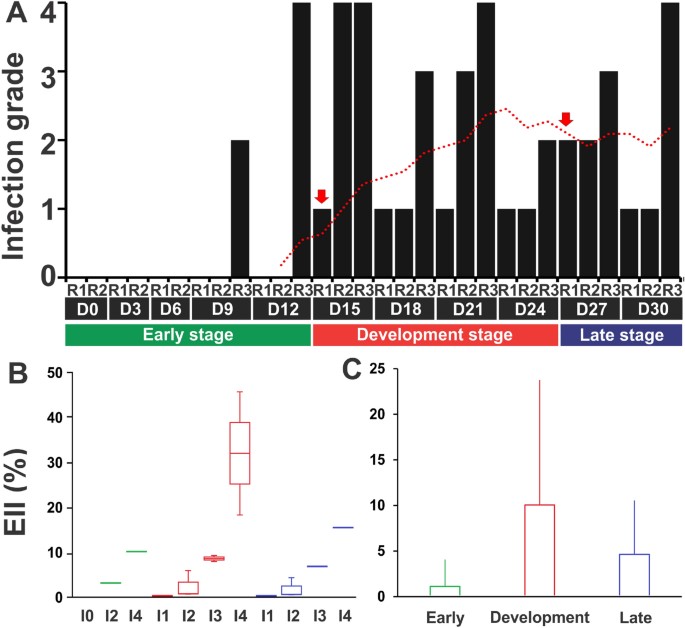
Hình 3: Đánh giá cấp độ nhiễm EHP. (A) Cấp độ nhiễm EHP (I0 – I4) được ghi nhận lại trong xét nghiệm sinh học và giá trị chỉ số nhiễm EHP (EII) được tính theo (B) mức độ nhiễm của từng giai đoạn bệnh và (C) giá trị trung bình cho từng giai đoạn bệnh. Các thanh màu đen biểu thị mức độ lây nhiễm của từng con được lấy mẫu. Đường chấm màu đỏ thể hiện giá trị trung bình của các thanh màu đen sau 3 ngày lấy mẫu. Các mũi tên màu đỏ thể hiện sự bắt đầu và kết thúc của mỗi giai đoạn bệnh. Các giai đoạn của bệnh được thể hiện qua các thanh màu, giai đoạn đầu (xanh lá), giai đoạn đang phát triển (đỏ) và giai đoạn cuối (xanh dương). R: bản sao (tôm lấy mẫu); D: ngày (ngày lấy mẫu). Giá trị EII được thể hiện dưới dạng trung bình ± độ lệch chuẩn.
Do sự biến đổi của quá trình lây nhiễm của EHP, nên không thể đảm bảo các bản sao cho từng cấp độ nhiễm cho mỗi giai đoạn. Vì lý do này, các số liệu thống kê về sự phát triển của bệnh EHP chỉ mang tính mô tả. Giai đoạn đầu bao gồm 12 ngày đầu tiên sau khi nhiễm bệnh (dpi) khi giá trị trung bình EII thấp hơn (0,92 ± 3,06%). EHP chỉ được phát hiện ở một con tôm được lấy mẫu trên 9 và 12 con sau khi nhiễm bệnh. Sau giai đoạn đầu, tất cả tôm được lấy mẫu đều dương tính với EHP (I1 – I4). Giai đoạn phát triển bắt đầu ở ngày 15 và kết thúc ở ngày 24 sau khi nhiễm, và giá trị trung bình của EII cao (8,08 ± 13,75%). Giai đoạn này được đặc trưng bởi sự gia tăng đột ngột của tôm nhiễm ở các cấp độ nhiễm cao (I3 và I4) và tôm ở cấp I4 có giá trị EII cao nhất (31,97 ± 19,37%) trong suốt quá trình thử nghiệm sinh học. Ở giai đoạn cuối (27-30 ngày sau khi nhiễm), số lượng tôm có cấp nhiễm cao (I3 và I4) giảm và giá trị trung bình EII giảm (5,44 ± 6,28%). Ở giai đoạn này, những gì chúng tôi quan sát thấy dường như là một mô hình tiệm cận nơi tôm đang trong quá trình nhiễm bệnh theo chu kỳ. Theo các kết quả này, EHP có thể phát triển cho đến khi tôm đạt mức nhiễm I4 và sau đó tải lượng ký sinh trùng bắt đầu giảm cho đến khi tôm trở lại mức nhiễm I1. Mô hình này có thể được lặp lại trong suốt chu kỳ nuôi.
Phân tích thành phần vi sinh vật
Đối với các phép phân loại vi khuẩn và nấm, tổng số 532,841 và 658,492 lần đọc trình tự không phải dạng số (Q> 25) đã được sử dụng để xây dựng bảng OTUs bằng cách sử dụng cơ sở dữ liệu Greengenes và UNITE. Tổng số 1895 OTU cho vi khuẩn và 213 OTU cho nấm đã được tìm thấy và được sử dụng làm tài liệu tham khảo cho các thư viện. Trong số các vi khuẩn, sự phân loại theo hệ thống cho phép xác định hai lĩnh vực, vi khuẩn (97-100%) và cổ khuẩn (<3%). Tổng cộng có 20 vi khuẩn (11 chưa phân loại), 3 vi khuẩn phyla cổ được xác định và phân loại thêm thành 234 (215 chi chưa được phân loại). Trong số các loài nấm, một miền đã được xác định là nấm. Tổng cộng có 3 loài nấm phyla đã được xác định và phân loại thêm thành 32 (5 chi chưa được phân loại).
Phyla tiêu biểu nhất được tìm thấy trong số các vi khuẩn bao gồm Proteobacteria (12–98%), Firmicutes (1–24%) và Tenericutes (0,3–82%) (Hình 4A) trong khi Basidiomycota (0–99%) và Ascomycota (0–99 %) có nhiều nhất trong số các loại nấm (Hình 4B). Sự khác biệt giữa các giai đoạn bệnh ở cấp độ phylum chỉ được thấy ở nấm, nơi mà Ascomycota phyla giảm và Basidiomycota phyla tăng ở giai đoạn cuối của bệnh.
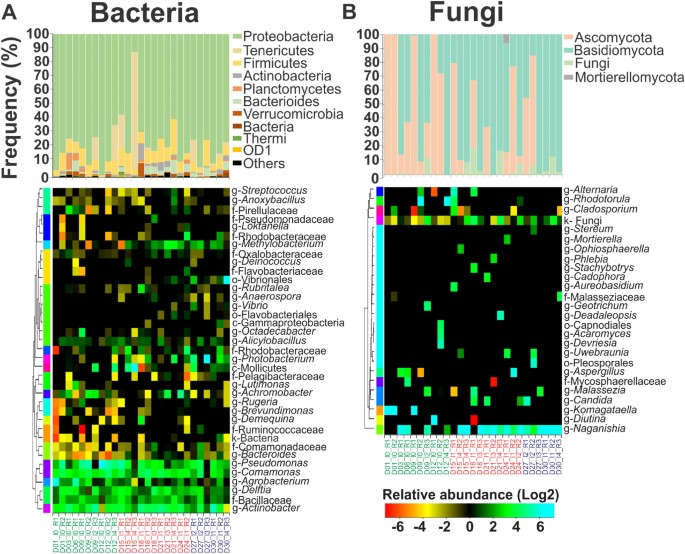
Hình 4: Hệ vi sinh vật tôm ở cấp phyla và chi được thể hiện ở các giai đoạn bệnh khác nhau. Điều chỉnh hệ vi sinh vật đối với đơn vị phân loại (A) vi khuẩn và (B) nấm. Mức độ phong phú tương đối được biểu thị bằng tỷ lệ phần trăm đối với phyla, trong đó chỉ phyla có mức độ phong phú tương đối cao hơn 1% đối với ít nhất một trong các lần lặp lại được hiển thị. Bản đồ nhiệt sử dụng mức độ phong phú tương đối được biến đổi Log2 được sử dụng để phân nhóm (trục y) các chi có mặt trong các điều kiện tương tự. Chỉ các chi có mức độ phong phú tương đối cao hơn 1% đối với ít nhất một trong các lần lặp lại mới được hiển thị. Tôm được phân nhóm (trục x) theo giai đoạn bệnh (đầu – màu xanh lục, phát triển – màu đỏ và cuối – màu xanh lam) để dễ hình dung về sự thay đổi hệ vi sinh vật giữa tôm khỏe mạnh và tôm nhiễm bệnh. Màu của bản đồ nhiệt có phạm vi từ màu cam đối với vi sinh vật có lượng vi sinh vật phong phú thấp (<1%) đến màu lục lam đối với vi sinh vật có lượng vi sinh vật phong phú cao (1–90%). Các chữ cái p- (phyla), c- (class), o- (order), f- (family) và g- (genera) ở phía bên trái của tên đơn vị phân loại đại diện cho cấp nhóm.
Ở vi khuẩn, chi xuất hiện nhiều nhất là Pseudomonas (0,7–80%) (Hình 4A) và ở nấm là Naganishia (0–97%) (Hình 4B). Ở vi khuẩn, Pseudomonas, Comamonas, Agrobacterium, Delftia và Actinobacteria được tập hợp riêng biệt với các chi khác như Streptococcus, Anoxybacillus, Loktanella, Methylobacterium và Vibrio (Hình 4A). Ở nấm, hầu hết các chi đều tập hợp chung với nhau, ngoại trừ Naganisha (Hình 4B). Việc phân nhóm này cho phép xác định các vi sinh vật có thể được tìm thấy trong một môi trường vi mô cụ thể (ví dụ: các thông số hóa lý và các giai đoạn sinh lý của vật chủ).
Theo Jesús Antonio López-Carvallo, Roberto Cruz-Flores & Arun K. Dhar
Biên dịch: Huyền Thoại – Tôm Giống Gia Hóa Bình Minh
“Tôm Giống Gia Hóa – Chìa Khóa Thành Công”
Xem thêm:
- Phần 2 – Tác Nhân Gây Bệnh Mới – Enterocytozoon hepatopenaei Làm Suy Giảm Hệ Vi Sinh Vật Theo Chu Kỳ Trong Gan Tụy Của Tôm Thẻ (Penaeus vannamei)
- Sự Đồng Nhiễm Virut Gây Bệnh Hoại Tử Cơ Và Enterocytozoon Hepatopenaei (EHP) Trong Các Trang Trại Thẻ Chân Trắng Ở Bờ Biển Phía Đông Của Ấn Độ
- Phần 1 – Nghiên Cứu Về Hoạt Động Kiếm Ăn Của Tôm Thẻ Chân Trắng Dựa Trên Phát Hiện Âm Thanh Thụ Động

 English
English