4/ Các phương pháp phát hiện EHP và định lượng
4.1 Thảo luận chung
Phát hiện EHP bằng kính hiển vi ánh sáng được đề cập trong Phần 3 của bệnh lý EHP nhưng không được khuyến khích cho các mục đích thông thường, đặc biệt là trong giai đoạn sống của tôm con và tôm lớn hơn sau này vì những lý do đã nêu trong đó. Các phương pháp phát hiện tế bào mô, đặc biệt là khuếch đại axit nucleic, thường có độ nhạy và độ đặc hiệu cao, và có thể cần ít thời gian chuẩn bị hơn so với yêu cầu về mô học. Phần này cung cấp một cái nhìn tổng quan về tất cả các phương pháp phân tử đã được phát triển cho EHP, cũng như các nền tảng đầy hứa hẹn nhưng chưa được áp dụng để phát hiện EHP. Các khía cạnh khác nhau của mỗi phương pháp, bao gồm cả độ nhạy, tính đặc hiệu, khả năng tiếp cận và định lượng, được tóm tắt và được xếp hạng trong Bảng 1. Đối với những người quan tâm đến việc triển khai các kỹ thuật công nghệ này, trình tự mồi và thăm dò từ các nghiên cứu trước đó cũng được đưa ra trong Bảng 2.
4.2 Chuẩn bị mẫu cho phương pháp phân tử.
Chúng tôi muốn lưu ý rằng việc lấy mẫu HP thích hợp là điều cần thiết để đảm bảo độ chính xác trong việc phát hiện và định lượng EHP, do sự phân bố không đồng đều của EHP bên trong HP theo quan sát của ISH (Flegel và Sritunyalucksana, 2018) (Hình 7, 10). Nếu chỉ một phần nhỏ HP bị cô lập thì vẫn có cơ hội rằng phần được lấy mẫu có thể chứa ít hơn hoặc nhiều hơn EHP so với trung bình cho toàn bộ cơ quan. Do đó, chúng tôi khuyên bạn nên chuẩn bị toàn bộ HP đồng nhất, sau đó loại bỏ một mẫu phụ cho chiết xuất mẫu DNA sẽ được sử dụng trong phân tích tiếp theo. Đây sẽ giúp ngăn ngừa sự xuất hiện của phủ định sai và đánh giá quá cao hoặc đánh giá thấp mức độ nghiêm trọng của nhiễm trùng. Vì lý do tương tự, chúng tôi cũng khuyến cáo thận trọng trong việc giải thích mức độ nghiêm trọng, sinh thiết kim HP được sử dụng để chuẩn bị mẫu DNA cho EHP qPCR (Cruz-Flores và cộng sự, 2019).
4.3 PCR
Phản ứng chuỗi polymerase (PCR) vẫn là tiêu chuẩn vàng để chẩn đoán nhiều bệnh tôm như được nêu trong các quy trình của Tổ chức Thú y Thế giới (OIE, 2019a, 2019b, 2019c). Một số các phương pháp dựa trên PCR đã được mô tả để phát hiện EHP, bao gồm PCR một bước (Tang và cs., 2015; Tourtip cs., 2009), nested-PCR (Jaroenlak và cs, 2016; Tangprasittipap và cs., 2013), và multiplex PCR có thể phát hiện EHP cùng với các mầm bệnh tôm khác (Koiwai và cộng sự, 2018).
Nói chung, PCR một bước đơn giản hơn để thực hiện và chỉ yêu cầu một bộ mồi, nhưng giới hạn phát hiện thường nằm trong khoảng từ 1000–10.000 bản sao cho mỗi phản ứng, không đủ nhạy để phát hiện những nhiễm trùng không có triệu chứng. Nested – PCR sử dụng hai bộ mồi để khuếch đại mục tiêu theo trình tự, mang lại độ nhạy cao hơn ít nhất 10 lần so với PCR một bước, và do đó cung cấp khả năng phát hiện nhiễm trùng mức độ thấp. Các hướng dẫn về nested-PCR được phát triển cho EHP có giới hạn phát hiện 10 bản sao cho mỗi phản ứng (Jaroenlak và cộng sự, 2016; Tang prasittipap và cộng sự, 2013). Một lợi ích khác của các phương pháp nested- PCR là bán định lượng. Nhiễm trùng nặng sẽ phát sinh hai amplicon từ vòng khuếch đại đầu tiên và thứ hai, trong khi nhiễm ánh nhẹ chỉ mang lại một amplicon trong bước PCR thứ hai (Jaroenlak và cộng sự, 2016).
Mặc dù các phương pháp dựa trên PCR thường đủ cụ thể để xác định các loài, nhưng việc hiểu sai có thể xảy ra nếu gen được chọn từ các loài mục tiêu thể hiện sự tương đồng cao với các loài từ các sinh vật có quan hệ họ hàng gần khác. Các gen SSU-rRNA đã được sử dụng rộng rãi làm PCR mục tiêu để xác định loài và chẩn đoán mầm bệnh vì trình tự thường đủ khác nhau giữa các loài khác nhau trong khi khá giống nhau trong cùng một loài. Vì lý do này, các chẩn đoán ban đầu dựa trên PCR được báo cáo cho EHP được thiết kế để nhắm mục tiêu vào gen SSU rRNA (Koiwai và cộng sự, 2018; Tang và cộng sự, 2015; Tangprasittipap và cộng sự, 2013; Tourtip và cộng sự, 2009). Tuy nhiên, nhiều trình tự sắp xếp sau đó đã tiết lộ danh tính trình tự cao giữa các SSU-rRNA đồng đẳng từ các vi bào tử trùng dưới nước tại các điểm gắn mồi của các đoạn mồi (68–90% độ đồng nhất) (Jaroenlak và cộng sự, 2016). Thật vậy, mồi dựa trên gen SSU-rRNA từ EHP cho thấy phản ứng chéo đáng kể với các vi sinh vật dưới nước có liên quan chặt chẽ như Enterospora canceri (Stentiford và cs, 2007) và Hepatospora eriocheir (Jar oenlak và cs, 2016; Stentiford và cs, 2011). Do đó, mồi SSU-rRNA có thể không phù hợp để kiểm tra phân, thức ăn và nước ao nuôi tôm trong đó các vi bào tử trùng liên quan khác có thể hiện diện.
Thay cho gen SSU-rRNA, Jaroenlak và cs (2016) đã thiết kế một phương pháp nested-PCR nhắm mục tiêu gen protein thành bào tử (EhSWP1), có trình tự là duy nhất cho EHP. Nested PCR, cụ thể cho EhSWP1 này giao thức (SWP-PCR) có thể phân biệt thành công EHP với các vi bào tử trùng liên quan chặt chẽ khác, đồng thời hiển thị độ nhạy cao (10 bản sao cho mỗi phản ứng). Để đảm bảo tính đặc hiệu phân biệt, chúng tôi khuyến cáo rằng các ứng dụng PCR trong tương lai được thiết kế xung quanh gen EhSWP1 hoặc các gen khác đa dạng về trình tự cao giữa các vi bào tử trùng dưới nước được sử dụng, đặc biệt trong thức ăn, phân và môi trường mẫu.
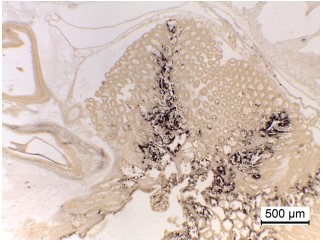
Hình 10. Hình ảnh với độ phóng đại thấp của mô HP bị nhiễm EHP từ xét nghiệm ISH với mẫu tôm con. Mẫu vật có tương đối ít tế bào đang tạo ra bào tử được xác định bằng phân tích mô học thông thường sử dụng nhuộm H&E thông thường. Mặc dù vậy, một tỷ lệ cao của ống các tế bào biểu mô ở khu vực trung tâm và trung gian của HP có thể được nhìn thấy là dương tính (nhuộm màu tối) cho thấy sự hiện diện của EHP, trong khi khu vực tế bào E xa vùng trung tâm cho kết quả nhuộm âm tính. Ngoài ra, sự phân bố của nhiễm các tế bào trong HP không đồng đều, do đó các mẫu nhỏ được lấy ngẫu nhiên từ các vùng của HP có thể cho kết quả PCR định lượng rất khác nhau đối với mức độ nghiêm trọng của nhiễm trùng EHP.
Bảng 1. So sánh độ nhạy cảm, đặc hiệu, khả năng tiếp cận và khả năng định lượng của các phương pháp chẩn đoán. Mỗi phương pháp được xếp loại từ A-D (A: tốt nhất, D: tệ nhất).
|
Phương pháp |
Độ nhạy cảm | Độ đặc hiệu | Khả năng tiếp cận | Khả năng định lượng |
Dẫn chứng |
| Các phương pháp chẩn đoán lâm sàng | |||||
| Sử dụng kính hiển vi ánh sáng truyền qua | D | C | B | C | Tourtip và cs. (2009) Sanguanrut và cs. (2018) |
| Lai tại chỗ ISH | C | B | D | B | Tangprasittipap và cs. (2013) Tang và cs. (2015) |
| Các phương pháp phân tử | |||||
| PCR một bước | B | B (SSU-rRNA) | C | C | Tourtip và cs. (2009)
Tang và cs. (2015) Koiwai và cs. (2018) |
| Nested PCR | A | B (SSU-rRNA)
A (SWP) |
C | B | Tangprasittipap và cs. (2013)
Jarroenlak và cs (2016) |
| LAMP | A | B (SSU-rRNA) | A | C | Suebsing và cs. (2013)
Sathish Kumar và cs. (2018) |
| qPCR | A | B (SSU-rRNA)
|
D | A | Liu và cs. (2016)
Liu và cs. (2018) |
| RPA | A | B (SSU-rRNA)
A (PTP2) |
A | C | Kanitchinda và cs. (2020)
Zhou và cs. (2020) |
Bảng 2. Các mồi được sử dụng trong các phương pháp phân tử để phát hiện EHP
| Phương pháp | Dẫn chứng | Gen đích | Tên | Đoạn mã (5’-3’) | Ghi chú |
| PCR một bước | Tourtip và cs. (2009) | SSU-rRNA | MF1
MR1 |
CCGGAGAGGGAGCCTGAGA
GACGGGCGGTGTGTACAAA |
|
| Tang và cs. (2015) | SSU-rRNA | EHP-510F
EHP-510R |
GCCTGAGAGATG GCTCCCACGT
GCGTACTATCCCCAGAGCCCGA |
||
| Nested PCR | Tangprasittipap và cs. (2013)
|
SSU-rRNA | ENF779 | CAGCAGGCGCGAAAATTGTCCA | Mồi bước đầu tiên |
| ENF779 | AAGAGATATTGTATTGCGCTTGCTG | Mồi bước thứ 2 | |||
| ENF176 | CAACGCGGGAAAACTTACCA | ||||
| ENF176 | ACCTGTTATTGCCTTCTCCCTCC | ||||
| Jarroenlak và cs (2016) | EhSWP | SWP_1F | TTGCAGAGTGTTGTTAAGGGTTT | Mồi bước đầu tiên | |
| SWP_1R | CACGATGTGTCTTTGCAATTTTC | Mồi bước thứ 2 | |||
| SWP_2F | TTGGCGGCACAATTCTCAAACA | ||||
| SWP_2R | GCTGTTTGTCTCCAACTGTATTTGA | ||||
| LAMP | Suebsing và cs. (2013) | SSU-rRNA | F3 | GATGCTTGGTGTGGGAGAA | |
| B3 | CCCCCCATCAATTTCCAACG | ||||
| FIP | TCCTGGTAGTGTCCTTCCGTCA/TTTT/TTTTCGGGCTCTGGGGATA | ||||
| BIP | AACGCGGGAAAACTTACCAGGG/TTTT/ GCACCACTCTTGTCTACCTC | ||||
| LF | TCACCCTTGCGAGCGT | ||||
| BF | TCAAGTCTATCGTAGATTGGAGACA | ||||
| Thiol probe | TCAAGTCTATCGTAGATTGGAGACA | SH = Nhóm thiol | |||
| Sathish Kumar và cs. (2018) | SSU-rRNA | F3 | TGGAGGGCAAGTTTTGGTG | ||
| B3 | GAGCATCGCTTTCGCCTC | ||||
| FIP | GAGCATCGCTTTCGCCTC | ||||
| BIP | AGTAGCGGAACGGATAGGGAGC/TTTT/CCAGGTGGGGTCTTGAG | ||||
| LF | ACTGCAGCATCCACCATA | ||||
| BF | ATGGTATAGGTGGGCAAAGAATG | ||||
| qPCR | Liu và cs. (2016) | SSU-rRNA | ENF185 | GTAGCGGAACGGATAGGG | |
| ENR185 | CCAGCATTGTCGGCATAG | ||||
| Liu và cs (2018) | SSU-rRNA | F157 | AGTAAACTATGCCGACAA | ||
| R157 | AATTAAGCAGCACAATCC | ||||
| Taqman Probe | FAM-TCC TGG TAG TGT CCT TCC GT-TAMRA | FAM = fluorophore; TAMRA =
quencher |
|||
| RPA | Zhou và cs. (2020) | SSU-rRNA | F2 | CATTGAGTTTGTTGAGAGTAGCGGAACGGAT | |
| R2 | CTAAGAGCATCGCTTTCGCCTCCGTTGGTC | ||||
| Kanitchinda và cs. (2020)
|
PTP2 | EHP-F1 | CACTCAAGGAATGGCTCAAGGGTTCAAAAT | Đi kèm với phát hiện CRISPR-Cas12a | |
| EHP-R1 | ACTGCTACACAAAGGCAGCACTCAAGGAAT | ||||
| Guide RNA | GUAAUUUCUACUAAGUGUAGAUGAUGUGUUUAAGAACGUAGU |
4.4 Các phương pháp khuếch đại đẳng nhiệt: LAMP and RPA
Ngày càng có nhiều sự tập trung vào việc điều chỉnh dựa trên các phương pháp phát hiện trong phòng thí nghiệm để áp dụng tại ao, cho phép người nuôi tôm trực tiếp thực hiện giám sát một cách dễ dàng. Phương pháp khuếch đại DNA trong điều kiện đẳng nhiệt (LAMP) giúp nhiệt độ phản ứng không đổi (được cung cấp bởi bể điều nhiệt đơn giản), loại bỏ việc phải sử dụng máy quay nhiệt đắt tiền (Mori và Notomi, 2009). Ngoài ra, LAMP có thể nhạy hơn gấp 1000 lần PCR một bước, giúp phát hiện nhiễm trùng ở trạng thái mang ở tôm (Wong và cộng sự, 2017). Một số các phương pháp phát hiện dựa trên LAMP đã được mô tả cho mầm bệnh trên tôm bao gồm WSSV, YHV, IHHNV và Vibrio parahaemolyticus (Arunrut và cộng sự, 2016; Kono và cộng sự, 2004; Mekata và cộng sự, Năm 2006; Sun và cộng sự, 2006).
Để phát hiện EHP, Suebsing và cộng sự (2013) đã phát triển một nền tảng sử dụng LAMP kết hợp với nanogold liên hợp thăm dò (Suebsing và cs., 2013). Phương pháp này có thể phát hiện EHP với ít nhất 0,02 fg tổng số DNA từ mô bị nhiễm bệnh, tương đương với 1 bản sao EHP cho mỗi hỗn hợp phản ứng, nhạy hơn so với bản sao nested-PCR. Gần đây hơn, SYBR Green I, một loại thuốc nhuộm DNA huỳnh quang, được sử dụng để phát hiện trực quan các amplicon LAMP tích cực, với giới hạn phát hiện 10 bản sao DNA plasmid (Sathish Kumar và cộng sự, 2018). Tuy nhiên, vì tôi có thể xen kẽ SYBR Green với bất kỳ DNA nào mà không cần quan tâm đến trình tự (Zipper và cộng sự, 2004), kết quả phát hiện dương tính giả có thể xảy ra nếu có amplicon LAMP không cụ thể. Do đó, tất cả các ứng dụng LAMP được báo cáo cho EHP được thiết kế để nhắm mục tiêu gen SSU-rRNA của nó, nhưng yêu cầu đối với cặp mồi 4–6 có thể làm cho LAMP ít nhạy cảm hơn PCR thành dương tính giả do microsporidia có liên quan gần gây ra. Tuy nhiên, để giảm thiểu cơ hội phản ứng chéo, chúng tôi khuyên bạn nên dùng Gen EhSWP1 hoặc một mục tiêu tương tự, cụ thể hơn trong việc thiết kế LAMP mới để phát hiện mầm bệnh này.
Khuếch đại polymerase tái tổ hợp (RPA) là một quá trình kỹ thuật đẳng nhiệt khuếch đại có thể khuếch đại DNA ở nhiệt độ cố định từ 37 đến 42 ◦C, với thời gian phản ứng ngắn nhất là 10 phút (Piepen burg và cộng sự, 2006; Euler và cộng sự, 2012; Krolov ˜ và cộng sự, 2014). Không giống như LAMP, RPA chỉ yêu cầu 1 cặp mồi và tạo ra một amplicon duy nhất tương thích với các quy trình xuôi dòng như nhân bản và giải trình tự Sanger. Zhou và cộng sự. đã phát triển một phương pháp RPA để phát hiện Gen SSU-rRNA từ EHP với độ nhạy 800 bản sao cho mỗi phản ứng (Zhou và cộng sự, 2020). Tuy nhiên, kết quả được đánh giá chỉ với điện di trên gel, có thể không thuận tiện cho các ứng dụng “point-of-need” và độ đặc hiệu không được đánh giá so với các loại microsporidia thủy sinh khác. Gần đây, Kanitchinda và cs. đã báo cáo sự kết hợp của RPA với CRISPR-Cas12a để phát hiện EHP (Kanitchinda và cộng sự, 2020). Các mồi được thiết kế để nhắm mục tiêu gen protein vòi phân cực 2(ptp2), gen này cho thấy sự tương đồng về trình tự thấp giữa các loài microsporidian. Trong một mẫu dương tính, endonuclease Cas12a sẽ phân cắt amplicon RPA theo trình tự cụ thể, sau đó tiến hành phân cắt trình liên kết ssDNA trong fluorophore-quencher R, dẫn đến phát xạ huỳnh quang có thể được hình dung bằng cách tiếp xúc với tia UV hoặc ánh sáng xanh. Với một chút thay đổi, cũng có thể đọc kết quả với bên phát hiện dòng chảy, loại bỏ nhu cầu về thiết bị trực quan đắt tiền. Các giới hạn phát hiện của phương pháp này là 50 bản sao EHP cho mỗi phản ứng và không phản ứng chéo với các vi bào tử trùng thủy sinh có liên quan gần được quan sát. Mặc dù nền tảng này chưa được sắp xếp hợp lý để sử dụng bên ngoài phòng thí nghiệm, với sự tối ưu hóa hơn nữa, nó có thể trở nên thích hợp hơn cho việc giám sát thường xuyên tại chỗ về EHP.
4.5 Phương pháp định tính
Trong khi kết quả định tính, tất cả hoặc không có kết quả thường thích hợp cho việc quản lý bệnh, các phương pháp định lượng vẫn cần thiết để xác định mức độ nghiêm trọng của nhiễm bệnh và tiến triển của bệnh.
PCR định lượng thời gian thực (qPCR) là một tiêu chuẩn vàng để đánh giá lượng tử mầm bệnh dựa trên mối tương quan tuyến tính giữa logarit của số bản sao tiêu bản và số chu kỳ PCR cần thiết để đạt đến ngưỡng xác định trước (Kralik và Ricchi, 2017). Để phát hiện EHP, Các xét nghiệm qPCR dựa trên huỳnh quang SYBR-Green I và đầu dò TaqMan là có thể phát hiện tương ứng là 83 và 40 bản sao của ký sinh trùng (Liu và cộng sự, 2016; Liu và cộng sự, 2018), và chúng hiển thị các phạm vi động trải dài một số bậc độ lớn.
Thú vị là, những nghiên cứu này đã báo cáo một xu hướng (nghĩa là không tìm thấy ở tất cả các trang trại) cho mối tương quan nghịch giữa bản sao số lượng EHP và kích thước cơ thể tôm, bất cứ khi nào số bản sao tăng lên trên 103 DNA tổng số mỗi ng được chiết xuất từ HP (Liu và cộng sự, 2016; Liu và cộng sự, 2018). LAMP thời gian thực cũng đã được mô tả, có thể phát hiện tổng số DNA bị pha loãng xuống 10-6 lần từ tôm nhiễm EHP với tính đặc trưng cao (Cai và cộng sự, 2018). Mặc dù có độ nhạy cao và khả năng định lượng, nhược điểm chính của qPCR và các biến thể của nó là yêu cầu của một máy qPCR đắt tiền, xử lý chất lỏng đáng kể và nhân viên được đào tạo chuyên sâu về sinh học phân tử. Sự phát triển của các thiết bị qPCR cầm tay giá rẻ, chẳng hạn như các thiết bị in 3D (Mendoza Gallegos và cộng sự, 2018), có thể làm cho kỹ thuật này hiệu quả về chi phí và thực tế trong cài đặt giới hạn tài nguyên.
4.6 Các phương pháp phân tử có triển vọng
Ngoài các phương pháp được mô tả ở trên, nhiều công cụ chẩn đoán hứa hẹn khác có sẵn nhưng vẫn chưa được điều chỉnh để phát hiện EHP, bao gồm cảm biến sinh học và giải trình tự thế hệ tiếp theo.
Cảm biến sinh học là thiết bị phát hiện có chứa nhận dạng sinh học phần tử được ghép nối với một bộ chuyển đổi hoặc bộ báo hóa lý (Ly Sin và cs., 2014), có thể được thiết kế để phát hiện nhiều loại chất phân tích khác nhau bao gồm DNA / RNA, protein, chất chuyển hóa, v.v. Ví dụ: cảm biến miễn dịch điện hóa dựa trên methylene blue-graphene oxide có thể phát hiện ít nhất 1,36×10-3 bản sao / μL của WSSV (Natarajan và cộng sự, 2017). Mặc dù những cảm biến sinh học này đặc biệt nhạy và định lượng, nhưng cũng có thể chịu chi phí nguyên vật liệu cao, độ nhạy đến ma trận sinh học, và sự phức tạp trong diễn giải dữ liệu (Ly Sin và cộng sự, 2014). Những hạn chế này sẽ cần được giải quyết trước khi chúng có thể đưa từ phòng thí nghiệm ra thực tế.
Giải trình tự gen thế hệ mới là một phương pháp giải trình tự nucleotide thông lượng cao có khả năng phát hiện một số lượng lớn mầm bệnh ngay lập tức mà không cần kiến thức tiên nghiệm, giúp bạn có thể khảo sát ngay cả các mầm bệnh chưa được biết đến trước đây (Al Rwahnih và cộng sự, 2015; Frey và cộng sự, 2014). Nó cũng có thể được sử dụng để theo dõi những thay đổi trong hệ vi sinh vật cả bên trong động vật chủ và trong môi trường để sử dụng làm chỉ thị hoặc dự đoán tình trạng sức khỏe động vật (Bass và cộng sự, 2019; Kim và Isaacson, 2015).
Một nghiên cứu trước đó báo cáo rằng một microsporidian có thể làm thay đổi hệ sinh thái vi sinh vật bên trong ruột sau của châu chấu, dẫn đến những thay đổi về hành vi và sinh lý, nhưng vẫn chưa biết liệu EHP có thể tác động như vậy trên tôm hay không (Tan và cs., 2015).
Do đó, giải trình tự gen thế hệ mới có thể cung cấp một cách để theo dõi những thay đổi trong hệ vi sinh vật trong phản ứng với EHP trong quá trình lây nhiễm, để cải thiện tình trạng bệnh sinh của EHP ngoài các tương tác trực tiếp giữa vật chủ và EHP.
Trong khi các giải trình tự gen thế hệ mới được biết là có chi phí thấp và đòi hỏi kỹ thuật cao, một dạng giải trình tự đĩa DNA / RNA trực tiếp di động, MinION, cũng như các biến thể của nó gần đây đã được thương mại hóa với mức giá rất phải chăng ($ 1000 cho thiết bị và bộ khởi động) và với đường ống phân tích dữ liệu thân thiện với người dùng (Brown và Clarke, 2016; Kono và Arakawa, 2019). Mặc dù có tiềm năng, những hạn chế chính của MinION bao gồm tốn thời gian chuẩn bị thư viện, độ nhạy thấp, khả năng thay đổi giữa các lần chạy cao và tỷ lệ lỗi cao (Tyler và cộng sự, 2018). Tuy nhiên, vì giải trình tự gen thế hệ mới là một lĩnh vực phát triển nhanh chóng, rất có thể nền tảng này có thể được cải thiện để khắc phục những nhược điểm này trong tương lai gần.
5/ Những nghiên cứu đang được thực hiện về các biện pháp kiểm soát
5.1 Thảo luận chung
Như các biện pháp phòng ngừa chống lại EHP, người nuôi tôm được khuyến cáo đảm bảo rằng nguồn cung cấp tôm giống (PL) âm tính với PCR và thực hiện các biện pháp an toàn sinh học thích hợp trước và sau khi thả giống để ngăn ngừa nhiễm từ các nguồn từ môi trường. Tuy nhiên, một khi EHP xâm nhập vào vì bất kỳ lý do gì, có thể rất khó để giảm thiểu tình trạng do hiện tại thiếu các phương pháp điều trị hiệu quả hoặc thiếu các biện pháp để kiểm soát HPM được chấp nhận. Do đó, các chiến lược kiểm soát thực tế tại trang trại là vẫn cần có và chúng tôi đề xuất rằng nên dựa trên quan sát và thông tin nghiên cứu phân tử. Phần này tóm tắt hiện tại và các hướng nghiên cứu tiềm năng có thể tạo điều kiện cho sự phát triển của các chiến lược kiểm soát đối với EHP.
5.2 Ức chế “sự đẩy” bào tử
Vì nhiễm EHP liên quan đến hoạt động “đẩy” của bào tử, tác động trực tiếp đến hoạt động này của bào tử EHP cũng là một chiến lược kiểm soát hiệu quả. Ví dụ, sử dụng phương pháp tinh sạch các bào tử nảy mầm in vitro cho thấy giảm khả năng lây nhiễm cao khi so sánh với bào tử không “đẩy” (Aldama-Cano và cộng sự, chưa xuất bản). Kết quả chỉ ra rằng ngoài việc khử trùng, ức chế vòi phân cực, sẽ có thể là phương pháp tiếp cận hướng tới việc vô hiệu hóa các bào tử EHP, đặc biệt là trong các ao hoặc trại giống có tiền sử nhiễm EHP trước đây.
Ảnh hưởng của pH đối với sự nảy mầm của bào tử cũng đã được chứng minh (Hình 11). Khi bào tử nguyên chất được ủ trong hệ đệm ở pH 4, 7 và 9, độ nảy mầm là 5, 10 và 90%, tương ứng, cho thấy rằng pH cao thúc đẩy bào tử đẩy ra. Vì vậy, việc sử dụng vôi sống (CaO) đã được khuyến khích để xử lý đáy trong quá trình chuẩn bị ao nuôi tôm để kích thích bào tử đẩy (tức là ngưng hoạt động) trước khi thả giống, đặc biệt là đối với các ao nuôi đã từng bị nhiễm trùng EHP.
Cuối cùng, nhiệt độ khắc nghiệt đã được chứng minh là có khả năng giết chết EHP (Aldama-Cano và cs., 2018). Chúng tôi khuyến cáo thức ăn tươi sống cho tôm như polychaetes và hai mảnh vỏ nên được đông lạnh ở – 20◦C trong ít nhất 48 giờ trước khi chúng được cho tôm bố mẹ ăn. Tuy nhiên, một phương pháp điều trị tốt hơn nhiều sẽ là giữ chúng ở 70◦C trong 15 phút (tiệt trùng) vì điều này sẽ không giết chết chỉ EHP mà còn các mầm bệnh vi khuẩn và vi rút chính khác của tôm, nếu có. Có thể là quá trình tiệt trùng sẽ làm giảm năng suất nauplii từ tôm bố mẹ, nhưng chúng tôi tin rằng chi phí này là hợp lý để loại bỏ nguy cơ ô nhiễm mầm bệnh cao từ các nguồn như vậy.

Hình 11. Biểu đồ thể hiện EHP được tinh sạch ở các chất đệm pH 4,7 và 9. Ghi chú tỉ lệ đẩy cao nhất ở pH 9.
5.3 Sự phát triển của các môi trường tế bào EHP
Nghiên cứu về cơ chế tăng sinh của EHP và tương tác với các vật chủ của nó được yêu cầu khẩn cấp nếu các chiến lược kiểm soát và phòng ngừa được phát triển. Thử nghiệm với các mô hình in vivo đã giúp cải thiện kiến thức của chúng tôi trong những lĩnh vực này, nhưng vẫn hạn chế, chẳng hạn như chi phí thử nghiệm, khả năng tương thích vật chủ-ký sinh trùng và sự can thiệp từ vật chủ đáp ứng miễn dịch hạn chế tốc độ của tiến trình nghiên cứu.
Mô hình in vitro không bị ràng buộc bởi những dòng như vậy sẽ đẩy nhanh tiến độ. Nỗ lực đầu tiên để phát triển các vi bào tử trong ống nghiệm là việc nuôi cấy Nosema bomycis trong các tế bào lót ống buồng trứng của tằm được thực hiện vào những năm 1930 (Franzen, 2008). Kể từ đó, một số microsporidians ký sinh cá, côn trùng và động vật trên cạn đã được nuôi cấy thành công ở ống nghiệm (Molestina và cộng sự, 2014). Ví dụ: các tế bào chính bắt nguồn từ bạch cầu cá cho phép Nucleospora salmonis phát triển từ Meronts sớm để thành bào tử trưởng thành và nó có thể được cấy truyền tới 17 lần bằng cách bổ sung định kỳ bạch cầu tươi từ cá khỏe mạnh vào các nền nuôi cấy ban đầu (Monaghan và cộng sự, 2009).
Một mô hình nuôi cấy tế bào dựa trên dòng tế bào lepidopteran đã được thiết lập cho N. ceranae và N. apis (ký sinh trùng của Ong mật phương Tây) và đã cho phép điều tra chi tiết về toàn bộ vòng đời trong điều kiện phòng thí nghiệm được kiểm soát cao. Nó dẫn đến việc quan sát thấy các mặt phẳng hình trục chính không xác định được mà trước đây đã bị bỏ qua ở những con ong bị nhiễm bệnh tự nhiên (Gisder và cộng sự, 2011).
Ngược lại, không có mô hình in vitro nào được phát triển cho động vật giáp xác bởi vì chưa có dòng tế bào bất tử nào được phát triển từ bất kỳ loài giáp xác nào. Một số nghiên cứu đã báo cáo việc sử dụng các tế bào nuôi cấy sơ cấp để nghiên cứu virus tôm (xem số đặc biệt của Crane và Benzie, 1999 về nghiên cứu nuôi cấy tế bào động vật không xương sống cho một lịch sử tổng quát). Các ví dụ bao gồm việc sử dụng các tế bào chính được phân lập từ mô tạo máu, bạch cầu, cơ quan lympho, tim, buồng trứng và tinh hoàn của tôm càng xanh, tôm càng xanh và cua để nghiên cứu WSSV (Jose và cộng sự, 2010; Li và cộng sự, 2015; Deepika và cộng sự, 2014; Jiang và cộng sự, 2006; Jiravanichpaisal và cộng sự, 2006; Jose và cộng sự, 2012; Li và cộng sự, 2014; Maeda và cộng sự, 2004; Nambi và cộng sự, 2012; Xu và cộng sự, 2018). Tuy nhiên, không có các nghiên cứu báo cáo về EHP, một phần là do việc thành lập nuôi cấy tế bào sơ cấp từ mô gan tụy của tôm đã được chứng minh là khó khăn hơn nhiều do ô nhiễm. Một vài nghiên cứu cho đến nay đã mô tả việc sử dụng thành công các mẫu nuôi cấy tế bào gan tụy sơ cấp, đó sẽ là những ứng cử viên tốt cho nghiên cứu về mô hình in vitro cho EHP (George và Dhar, 2010; Ghosh và cộng sự, 1995). Ngoài ra, dòng tế bào côn trùng có thể thay thế. Ví dụ, nó đã được báo cáo rằng Glugea sp. (microsporidian trên cá) có thể lây nhiễm sang tế bào côn trùng, nhưng sự khác biệt trong môi trường tế bào chủ vi mô có thể phức tạp hóa việc giải thích tương tác giữa mầm bệnh và vật chủ (Lores và cộng sự, 2003). Việc tìm kiếm (các) vật chủ có thể thay thế cho EHP có thể tiết lộ một phương pháp phù hợp hơn với nuôi cấy tế bào.
6/ Kết luận và những xu hướng nghiên cứu trong tương lai
6.1 Những kết luận chung và các nghiên cứu trong tương lai
Kể từ khi mô tả chính thức vào năm 2009 (mặc dù đã biết trước đó), EHP đã nổi lên là một trong những mầm bệnh quan trọng nhất ở châu Á đối với Tôm thẻ chân trắng kỳ lạ, được nuôi trồng Penaeus vannamei mà nó gây ra bệnh công khai HPM. Điều này trái ngược với lịch sử trước đây của nó thường Các bệnh nhiễm trùng ở mức độ thấp, khó hiểu trên tôm sú nuôi P. monodon. Liệu sự thay đổi về tác động tiêu cực của EHP có phải do vật chủ hay không chuyển đổi hoặc có sự khác biệt trong phương pháp canh tác (ví dụ: tăng mật độ nuôi với P. vannamei) vẫn còn là một câu hỏi. Trong bất kỳ trường hợp nào, nó có thể gây nhiễm trùng nặng dẫn đến chậm phát triển và yếu có thể dẫn đến nhiễm trùng thứ phát do cơ hội mầm bệnh. Cũng như các bệnh tôm lớn khác, việc phòng ngừa vẫn là tuyến phòng thủ tốt nhất chống lại EHP, đặc biệt là do tiết kiệm chi phí liệu pháp ở quy mô trang trại không có sẵn.
6.2 Tóm tắt những biện pháp được khuyến cáo hiện nay.
Rút ra từ những điểm có liên quan đã được thảo luận trong bài viết này, chúng tôi tóm tắt lại các nguyên tắc sau, nếu được tuân thủ đúng, có thể làm giảm đáng kể khả năng nhiễm EHP ở tôm trong hệ thống nuôi trồng:
- Nên tránh thức ăn tươi sống, đặc biệt là đối với tôm bố mẹ, hoàn toàn hoặc nếu phải sử dụng, trước tiên phải được tiệt trùng (điều kiện lý tưởng) hoặc đông lạnh ở -20◦C trong 48 giờ (ít lý tưởng hơn) để tiêu diệt các giai đoạn lây nhiễm của EHP.
- Nên bỏ đói tôm bố mẹ trong 4–6 giờ trước khi chuyển sang bể cho đẻ để giảm lượng phân bài tiết của chúng.
- Trứng hoặc nauplii trước giai đoạn N6 nên được rửa kỹ bằng nước sạch trong trại giống để loại bỏ các giai đoạn sống của EHP có thể có trong phân của con bố mẹ trong bể đẻ.
- Trước khi thả sang các ao (đặc biệt là những ao trước đây đã bị ảnh hưởng bởi EHP), nên xử lý bằng vôi sống (CaO) để kích thích sự nẩy mầm và bất hoạt bào tử để giảm lây nhiễm.
- PL cần được sàng lọc PCR để xác nhận không có EHP trước khi thả nuôi, cho dù chúng có được xác nhận là có mang mầm bệnh EHP hay không (SPF).
- Sau khi thả tôm giống âm tính với EHP, tôm trong ao cần được lấy mẫu định kì và xét nghiệm EHP bằng các phương pháp phân tử.
- Nếu kết quả lấy mẫu ao định kỳ dẫn đến phát hiện sớm EHP hoặc nếu HPM nghi ngờ, các biện pháp quản lý ao có thể được thay đổi để giảm tình trạng lây lan theo chiều ngang (ví dụ: giảm mật độ tôm, tăng cường trao đổi nước, loại bỏ phân, loại bỏ và chôn tôm chết hoặc tôm lờ đờ để ngăn ngừa ăn thịt đồng loại).
- Người nuôi tôm được khuyến khích đánh giá khả năng biến dị trong tính nhạy cảm di truyền với nhiễm EHP.
Nông dân nên tìm kiếm sự hỗ trợ của các chuyên gia kỹ thuật thích hợp để hỗ trợ giám sát định kỳ bằng cách sử dụng các công cụ chẩn đoán thích hợp và các công nghệ giải trình tự để phân biệt EHP với các vi bào tử EGM(và không phải EGM) khác.
Nhóm tác giả: Thawatchai Chaijarasphong a,b, Natthinee Munkongwongsiri c, Grant D. Stentiford d,e, Diva J. Aldama-Cano a,c, Kwanta Thansa c, Timothy W. Flegel a,f, Kallaya Sritunyalucksana c, Ornchuma Itsathitphaisarn a,g,*
Biên dịch: Thùy Linh – Công ty TNHH PTTS Bình Minh
“Tôm Giống Gia Hóa – Chìa Khóa Thành Công”
Xem thêm:
- Phần 1 – BỆNH DO VI BÀO TỬ TRÙNG Enterocytozoon hepatopenaei (EHP) GÂY RA TRÊN TÔM: Sinh thái, Bệnh học, Chẩn đoán và Kiểm soát
- Phần 2 – BỆNH DO VI BÀO TỬ TRÙNG Enterocytozoon hepatopenaei (EHP) GÂY RA TRÊN TÔM: Sinh thái, Bệnh học, Chẩn đoán và Kiểm soát
- ASC công bố tiêu chuẩn mới cho tôm nuôi trong hệ thống RAS

 English
English