Tóm tắt
Ngày càng có nhiều bằng chứng cho thấy hệ vi khuẩn đường ruột của tôm có liên quan chặt chẽ với các yếu tố môi trường, giai đoạn phát triển và tình trạng sức khỏe của vật chủ giống như ở người và động vật. Những nghiên cứu này đã cung cấp một hướng dẫn quan trọng để cải thiện lợi ích khi nuôi tôm. Trong thực tế, ngoài vi khuẩn, các vi sinh vật nhân chuẩn bị chi phối bởi hệ vi sinh vật nấm (Mycobiota), cũng đóng một vai trò quan trọng trong sự phát triển, trao đổi chất và cân bằng nội môi của vật chủ. Tuy nhiên, cho đến nay vẫn còn rất ít thông tin về hệ sinh vật nấm trong đường tiêu hóa của tôm. Nghiên cứu này đã sử dụng giải trình tự thông lượng cao của vùng đệm 1 được phiên mã nội bộ để mô tả đặc điểm của gan tụy và hệ sinh vật nấm đường ruột của tôm thẻ chân trắng và mối liên hệ của chúng với tỷ lệ mắc bệnh và sự thay đổi theo mùa. Kết quả cho thấy hệ sinh vật nấm đường ruột và gan tụy của tôm thẻ chân trắng Litopenaeus vannamei bị chi phối bởi ngành Ascomycota và Basidiomycota, và các chi Alternaria, Tuber, Hortaea, Sarocladium và Stagonospora. Hệ vi sinh vật nấm thay đổi đáng kể dưới ảnh hưởng của bệnh tật và sự thay đổi theo mùa. Tôm bị bệnh có mức độ nấm gây bệnh tiềm ẩn cao hơn, như chi Candida trong ruột. Tôm khỏe mạnh có nhiều chi Didymella và Filobasidium hơn trong ruột và Pyrenochaetopsis trong gan tụy. Đáng chú ý, hầu hết các loại nấm trên tôm thẻ chân trắng đều gây bệnh cho con người. Nghiên cứu này đã cho thấy hệ sinh vật nấm đường ruột và gan tụy của L. vannamei; cũng như tác động của bệnh và sự thay đổi theo mùa đối với hệ sinh vật nấm. Nghiên cứu này cung cấp hướng dẫn quan trọng cho việc nuôi tôm thẻ chân trắng ở Thái Bình Dương và cung cấp thông tin chi tiết hơn về hệ vi sinh vật nấm.
1. Giới thiệu
Hệ thống tiêu hóa chứa một số lượng lớn vi sinh vật do vi khuẩn chiếm ưu thế; tạo thành một hệ sinh thái khổng lồ được gọi chung là hệ vi sinh vật (O’Hara và Shanahan, 2006; Clemente và cộng sự, 2012). Hệ vi sinh vật có nhiều chức năng, chẳng hạn như duy trì sự ổn định chức năng và cân bằng trao đổi chất trong hệ thống tiêu hóa (Xue và cộng sự, 2015; Sommer và cộng sự, 2016; Sonnenburg và Backhed, 2016) và điều hòa phản ứng miễn dịch của vật chủ (Hooper và cộng sự, 2012; Palm và cộng sự, 2015). Do đó, nó rất quan trọng đối với vật chủ. Mặc dù hệ vi sinh vật của vi khuẩn chiếm ưu thế nhưng các vi sinh vật nhân chuẩn cũng xuất hiện (Neville và cộng sự, 2015; Sam và cộng sự, 2017). Nấm là hệ vi sinh vật nhân chuẩn quan trọng nhất (Neville và cộng sự, 2015). Gần đây, nhiều nghiên cứu đã chỉ ra rằng quần thể nấm đường ruột có mối tương quan chặt chẽ với các con đường điều hòa chuyển hóa và miễn dịch của vật chủ (Mar Rodriguez và cộng sự, 2015; McAleer và cộng sự, 2016; Huseyin và cộng sự, 2017; Sokol và cộng sự, 2017) và đang ngày càng nhận được nhiều sự chú ý.
Động vật giáp xác là phân ngành phong phú thứ hai trong Động vật chân đốt. Trong số này, tôm đã thu hút nhiều sự chú ý do giá trị thương mại quan trọng của chúng. Theo báo cáo, nuôi tôm có thể đạt sản lượng 3 triệu tấn mỗi năm (Zhang và cộng sự, 2016). Tôm thẻ chân trắng Litopenaeus vannamei là loài tôm phổ biến nhất, góp phần tạo nên những lợi thế sinh tồn như tốc độ tăng trưởng nhanh và khả năng thích nghi tốt với môi trường. Tuy nhiên, dịch bệnh xảy ra nhiều đã ảnh hưởng nặng nề đến nghề nuôi tôm. Các nghiên cứu gần đây cho thấy hệ vi khuẩn đường ruột của tôm thẻ chân trắng có liên quan chặt chẽ với tuổi vật chủ, tình trạng sức khỏe và phản ứng miễn dịch (Xiong và cộng sự, 2015; Cornejo-Granados và cộng sự, 2017). Hệ vi khuẩn đường ruột ổn định giúp tăng cường khả năng miễn dịch của tôm và khả năng chống lại mầm bệnh bên ngoài (Rungrassamee và cộng sự, 2016). Dựa trên kết quả, một phương pháp nuôi mới tổng hợp khả năng kháng bệnh của tôm bằng cách duy trì hoặc thiết lập lại hệ vi sinh vật đường ruột “khỏe mạnh” đã được phát triển và mang lại hiệu quả sâu rộng và tích cực cho nghề nuôi tôm (Xiong, 2018). Bên cạnh đó, một nghiên cứu về tôm thẻ chân trắng ở Thái Bình Dương đã cho thấy rằng cộng đồng vi khuẩn đường ruột thường xuyên thay đổi theo tiến trình và giai đoạn phát triển của bệnh tôm (Xiong và cộng sự, 2017). Những phát hiện mới đã dẫn đến việc một phương pháp chẩn đoán sớm chính xác hơn theo dõi hệ vi sinh vật đã xuất hiện và dần được áp dụng trong nuôi tôm (Xiong và cộng sự, 2017; Xiong, 2018).
Đặc biệt, một nghiên cứu gần đây đã nhắm mục tiêu vào gen 18S rRNA để phát hiện ra rằng hệ vi sinh vật nhân thực trong đường ruột của tôm chung sống ảnh hưởng đến quá trình tiêu hóa và hấp thụ dinh dưỡng và do đó làm thay đổi hiệu suất tăng trưởng (Dai và cộng sự, 2017). Tuy nhiên, cộng đồng nấm là quần thể có ảnh hưởng nhất trong hệ vi sinh vật nhân chuẩn đường ruột, vẫn chưa được khám phá rõ ràng do giải trình tự không đủ sâu trong nghiên cứu đó. Ngoài ra, một nghiên cứu dựa trên nuôi cấy cho thấy có nhiều loại nấm gây bệnh trong cơ thể tôm thẻ chân trắng (da Silva và cộng sự, 2011; Karthikeyan và cộng sự, 2015), có thể ảnh hưởng đến tình trạng sức khỏe của tôm nuôi. Tuy nhiên, số lượng nấm có thể nuôi cấy còn hạn chế (Huffnagle và Novrr, 2013), và sự tương tác sinh thái giữa các loài tồn tại bên trong đường tiêu hóa cũng hầu như không được phát hiện dựa trên phương pháp phụ thuộc vào nuôi cấy thông thường.
Ngoài ra, khác với hệ thống tiêu hóa của động vật có xương sống, hệ thống tiêu hóa của tôm là liên tục, bao gồm dạ dày, gan tụy và ruột. Trong số này, gan tụy đóng vai trò quan trọng trong việc điều chỉnh khả năng miễn dịch bẩm sinh của vật chủ, đồng thời nó cũng là cơ quan quan trọng chịu trách nhiệm cho quá trình tiêu hóa và hấp thụ (Cornejo-Granados và cộng sự, 2017). Tuy nhiên, cho đến nay, rất ít nghiên cứu tập trung vào hệ vi khuẩn trong các cơ quan tiêu hóa khác ngoài ruột, chưa kể đến hệ vi sinh vật nấm. Điều thú vị là một số nghiên cứu gần đây về vi khuẩn đường ruột của tôm cho thấy ngoài ruột, gan tụy cũng là nơi cư trú của nhiều loại vi khuẩn, có thể phản ứng với sự thay đổi của môi trường và tình trạng sức khỏe và có liên quan mật thiết đến khẩu phần ăn của vật chủ (Cheung và cộng sự, 2015; Cornejo-Granados và cộng sự, 2017). Tuy nhiên, sự biến đổi của hệ sinh vật nấm trong các phần khác nhau của đường tiêu hóa của tôm, chẳng hạn như gan tụy và ruột vẫn chưa được tiết lộ.
Nghiên cứu này đã sử dụng công nghệ giải trình tự thông lượng cao để phân tích hệ vi sinh vật nấm trong gan tụy và đường ruột của tôm thẻ chân trắng L. vannamei. Mục đích là để kiểm tra: (1) thành phần hệ vi sinh vật nấm đường ruột và gan tụy, (2) tác động theo mùa đối với thành phần hệ sinh vật nấm trong ruột và gan tụy, và (3) tác động của bệnh đối với thành phần hệ sinh vật nấm ở ruột và gan tụy của L. vannamei.
2. Vật liệu và phương pháp
2.1. Chuẩn mực đạo đức
Tất cả các phương pháp động vật thí nghiệm trong nghiên cứu này đã được phê duyệt bởi ủy ban về Đạo đức thí nghiệm động vật của Viện Tài nguyên sinh học ứng dụng Quảng Đông và tuân theo các nguyên tắc cơ bản.
2.2. Lấy mẫu
Vào mùa hè, căn cứ vào tình trạng dịch bệnh, chọn 3 ao nhiễm bệnh đại diện và 1 ao nuôi tôm khỏe mạnh. Vào mùa đông, chọn ngẫu nhiên 4 ao để lấy mẫu vì không tìm thấy tôm bệnh. Tất cả các bể nuôi được chọn trong nghiên cứu này đều sử dụng cùng một loại thức ăn nhân tạo (HAID GROUP, Trung Quốc). Tổng cộng có 50 con tôm thẻ chân trắng từ 8 trang trại đã được lấy mẫu, như thể hiện trong Bảng 1. Để tránh nhiễm bẩn, tôm sống được đánh bắt bằng lưới chài ngay lập tức được đặt trong các ống thu gom 50 mL đã khử trùng. Các ống thu gom được cho vào nước đá và nhanh chóng mang về phòng thí nghiệm địa phương. Mỗi con tôm được rửa kỹ bằng nước vô trùng và đặt vào đĩa vô trùng. Các mẫu đã rửa sạch được đặt trên trạm làm việc vô trùng để sinh thiết nhằm thu thập mô gan và mô ruột. Mô được lấy ra ngay lập tức được đặt trong các ống EP 2 mL và nhanh chóng được đặt trong đá khô. Các mẫu được bảo quản ở -80°C trong phòng thí nghiệm cho đến khi axit nucleic được chiết xuất.
Bảng 1. Mẫu tôm dùng để phát hiện nấm gan tụy và đường ruột.
2.3. Trích xuất DNA, khuếch đại PCR và giải trình tự
Đối với mỗi mẫu, tổng số DNA bộ gen được chiết xuất bằng cách sử dụng Bộ cách ly DNA PowerSoil® (MO BIO, Hoa Kỳ) theo hướng dẫn của nhà sản xuất. Sự phân lập DNA đã được định lượng bằng máy đo quang phổ ND-2000C (NanoDrop, Hoa Kỳ). Vùng đệm 1 được phiên mã nội bộ (ITS1) được khuếch đại bằng cách sử dụng bộ mồi phổ quát ITS1F 5′-CTTGGTCATTTAGAGGAAGTAA-3′ và ITS2 5′-GCTGCGTTCTTCATCGATGC-3′ (Mukherjee và cộng sự, 2014; Li và cộng sự, 2017). Phản ứng PCR được thực hiện bằng Phusion® Q5 DNA polymerase có độ trung thực cao (New England Biolabs, Vương quốc Anh) theo chương trình [95°C trong 5 phút, 15 × (95°C trong 30 giây, 50°C trong 30 giây, 72°C trong 40 giây), 72°C trong 7 phút, giữ ở 4°C; 98°C trong 30 phút, 15 × (98°C trong 30 giây, 65°C trong 30 giây, 72°C trong 30 giây), 72°C trong 5 phút, giữ ở 4°C]. Nước khử trùng được sử dụng làm đối chứng âm. Bộ mồi gắn các chuỗi mã vạch đã được sử dụng trong quá trình khuếch đại PCR thứ hai. Sau khi điện di trên gel agarose, các sản phẩm PCR được tinh chế bằng cách sử dụng Hạt sạch DNA VAHTSTM (Vazyme, Trung Quốc), sau đó là PCR Solexa, tinh chế hạt thứ hai. Các sản phẩm cuối cùng được gộp lại để tạo ra các thư viện giải trình tự. Các thư viện đã được giải trình tự trên nền tảng Illumina HiSeq (Illumina Hiseq 2500) bằng HiSeq Rapid Kit V2 (chu kỳ 500) (Illumina, Hoa Kỳ) theo hướng dẫn của nhà sản xuất.
2.4. Xử lý dữ liệu
Dựa trên các mã vạch duy nhất, chúng tôi đã chỉ định trình tự cho từng mẫu trước khi xóa mã vạch và trình tự mồi bằng QIIME v1.8.0 (Caporaso và cộng sự, 2010). FLASH v1.2.7 đã được sử dụng để thực hiện hợp nhất các lần đọc PE để thu được các chuỗi đã hợp nhất (thẻ thô) (Magoc và Salzberg, 2011). Độ dài tối thiểu của phần chồng lấp được đặt thành 10 bp và tỷ lệ không khớp tối đa trong vùng chồng lấp được đặt thành 0,2 (Magoc và Salzberg, 2011). Trimmomatic v 0.33 đã được áp dụng để tiến hành kiểm soát chất lượng tương đối nghiêm ngặt nhằm tạo ra một bộ thẻ sạch chất lượng cao (Bolger và cộng sự, 2014), tiếp theo là loại bỏ các trình tự khảm bằng UCHIME v 4.2 (Edgar và cộng sự, 2011) và trình tự không nấm với ITSx v 1.1.1 (Bengtsson-Palme và cộng sự, 2013). Sau đó, các thẻ hiệu quả chất lượng cao được nhóm lại để tạo ra các đơn vị phân loại vận hành (OTU) bằng cách sử dụng UCLUST v 1.2.22 với độ tương tự 97% (Edgar, 2013). Các OTU có độ phong phú thấp (số lượng trình tự dưới ba) đã được lọc và các OTU được chuẩn hóa đã được tạo (Bokulich và cộng sự, 2013). Theo cơ sở dữ liệu UNITE v 7.21 (Koljalg và cộng sự, 2013), trình tự đại diện của mỗi OTU được chú thích bằng RDP Classifier v 2.2 (Wang và cộng sự, 2007).
2.5. Phân tích thống kê
Các OTU được chuẩn hóa đã được sử dụng để vẽ biểu đồ thanh phân bố loài ở từng cấp phân loại, sau đó là xây dựng cây phát sinh loài bằng ClustalW22
Dữ liệu về độ phong phú được sử dụng để tính toán độ đa dạng α (các chỉ số đa dạng Chao 1, ACE, Richness, Shannon–Wiener và Simpson) bằng cách sử dụng Mothur v.1.3.03 (Schloss và cộng sự, 2009). Sự khác biệt giữa các nhóm được so sánh bằng phân tích phương sai một chiều (ANOVA). Chúng tôi đã vẽ đồ thị đường cong độ hiếm và đường cong độ phong phú tương đối của loài để đánh giá mức độ đầy đủ của độ sâu giải trình tự (Wang và cộng sự, 2012; Koljalg và cộng sự, 2013).
Độ đa dạng β của từng nhóm mẫu được tính toán dựa trên khoảng cách Bray–Curtis. Phân tích tọa độ chính đã được hoàn thành với gói R “ape” và một chương trình dendro phân cụm được xây dựng bằng phương pháp nhóm cặp không trọng số với trung bình số học (UPGMA). Sự khác biệt về đa dạng β giữa các nhóm được phát hiện bằng phân tích phương sai đa biến hoán vị (PERMANOVA).
Phân tích kích thước hiệu ứng phân tích phân biệt tuyến tính (LEfSe) được thực hiện ở từng cấp phân loại để sàng lọc các dấu hiệu phân tử (Dấu ấn sinh học) của từng nhóm (Segata và cộng sự, 2011).
2.6. Số gia nhập dữ liệu trình tự
Dữ liệu giải trình tự được tạo từ một trăm mẫu được mô tả trong nghiên cứu này có sẵn trong kho lưu trữ đọc trình tự (SRA) tại NCBI dưới số gia nhập PRJNA495902.
3. Kết quả
3.1. Đánh giá dữ liệu trình tự
100 mẫu đã được giải trình tự trên nền tảng Illumina Hiseq 2500 để tạo tập dữ liệu 12,56 Gb và 9.501.484 cặp lượt đọc. Trong số đó, 8.960.483 thẻ đã vượt qua kiểm soát chất lượng nghiêm ngặt và được xử lý thành 8.735.797 thẻ hiệu quả không có chimera chất lượng cao (trung bình là 87.358) (Bảng bổ trợ S1). Tổng cộng có 9.804 OTU được tạo với độ tương tự 97% (Bảng bổ trợ S2). Sau đó, các OTU, dưới ba trong tổng số thứ tự, đã được sàng lọc để thu được 8.491 OTU hiệu quả (Hình bổ sung S1). Các chỉ số đa dạng được tính toán dựa trên dữ liệu OTU. Các đường cong hiếm gặp và đường cong độ phong phú tương đối của loài đạt đến một cao nguyên, phản ánh sự đầy đủ của độ sâu trình tự (Hình bổ sung S2, S3).
3.2. Mô hình chung của hệ sinh vật nấm trong ruột và gan tụy của tôm thẻ chân trắng Litopenaeus vannamei
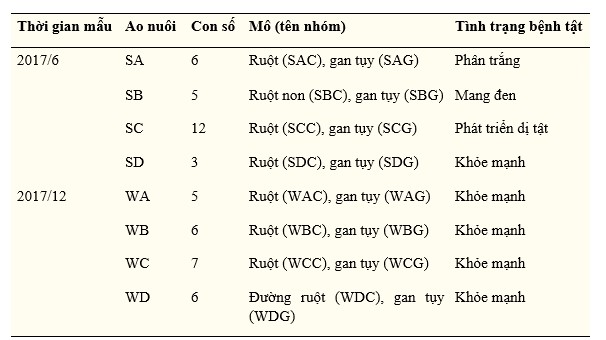
Tổng cộng có 17 ngành, 53 lớp, 127 bộ, 325 họ, 734 chi và 990 loài đã được xác định từ các trình tự có thể phân loại. Như được hiển thị trong Hình 1A, Ascomycota là ngành chiếm ưu thế nhất và chiếm 65,32% tổng số cộng đồng nấm, tiếp theo là Basidiomycota (12,45%). Các ngành khác với mức độ phong phú thấp là Mortierellomycota (1,19%), Chytridiomycota (0,21%) và Neocallimastigomycota (0,19%).
Hình 1. Phân bố loài của từng nhóm trong 50 con tôm. Bảng điều khiển (A) ở cấp độ ngành; bảng điều khiển (B) ở cấp chi (top 20=). ID mẫu được đề cập trong Bảng 1.
Ở cấp độ chi, 20 thành viên chiếm ưu thế hàng đầu là Alternaria (12,83%), Tuber (5,97%), Hortaea (4,93%), Sarocladium (3,95%), Stagonospora (3,47%), Saccharomyces (3,43%), Cladosporium (3,28%), Phaeophleospora (3,08%), Ramichloridium (1,37%), Mortierella (1,13%), Fusarium (1,05%), Auricularia (1,00%), Pleurotus (0,94%), Polyporus (0,93%), Pseudocercospora (0,86%), Trichosporon (0,84%), Aureobasidium (0,84%), Curvularia (0,74%), Malassezia (0,69%) và Candida (0,59%) (Hình 1B).
3.3. So sánh giữa hệ sinh vật nấm trong ruột và gan tụy
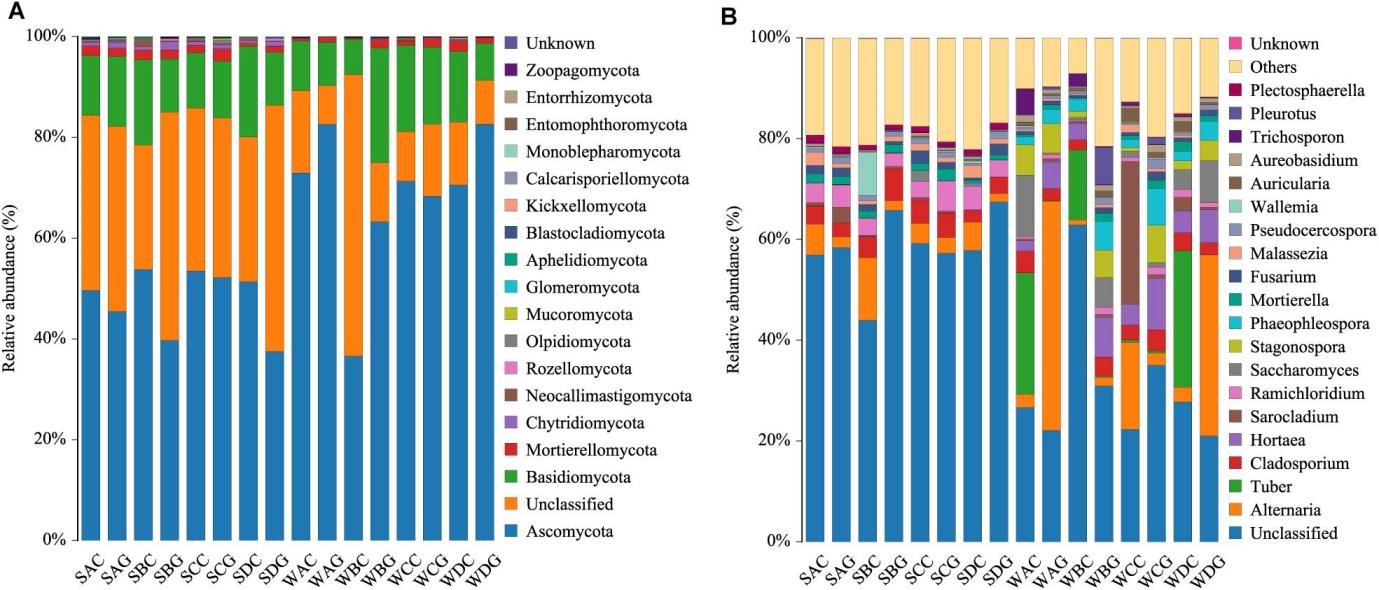
Một so sánh về sự đa dạng α đã được tiến hành giữa hệ vi sinh vật nấm đường ruột và gan tụy. Tầm quan trọng giữa các nhóm được phân tích bằng ANOVA. Để loại trừ sự can thiệp của các bệnh, khi phân tích sự khác biệt giữa các mẫu đường ruột và gan tụy mùa hè, chúng tôi chỉ chọn các mẫu lấy từ nhóm SD (nhóm đường ruột là SDC, nhóm gan tụy là SDG). Kết quả cho thấy rằng không phát hiện sự khác biệt đáng kể trong công cụ ước tính độ phong phú và công cụ ước tính độ đa dạng giữa nhóm đường ruột và gan tụy vào mùa hè (Bảng bổ trợ S3A). Đối với tính đa dạng β, mặc dù phân tích cụm UPGMA dường như cho thấy các mẫu mùa hè được chia thành 2 nhánh, cả kết quả PCoA và PERMANOVA đều cho thấy sự chồng chéo của hai nhóm. Điều này cho thấy sự tương đồng cao về thành phần cộng đồng và sự đa dạng giữa hai nhóm (Hình 2 và Bảng 2). Phân tích LEfSe đã được sử dụng để nắm bắt các loại nấm phân biệt giữa các nhóm. Basidiomycota, Pleosporales và Didymellacea cho thấy mức độ cao hơn trong nhóm đường ruột và Sordariomycetes được làm giàu đáng kể trong nhóm gan tụy. Tuy nhiên, không có loại nấm đại diện điển hình nào được tìm thấy ở cấp độ chi và loài (Hình 3A).
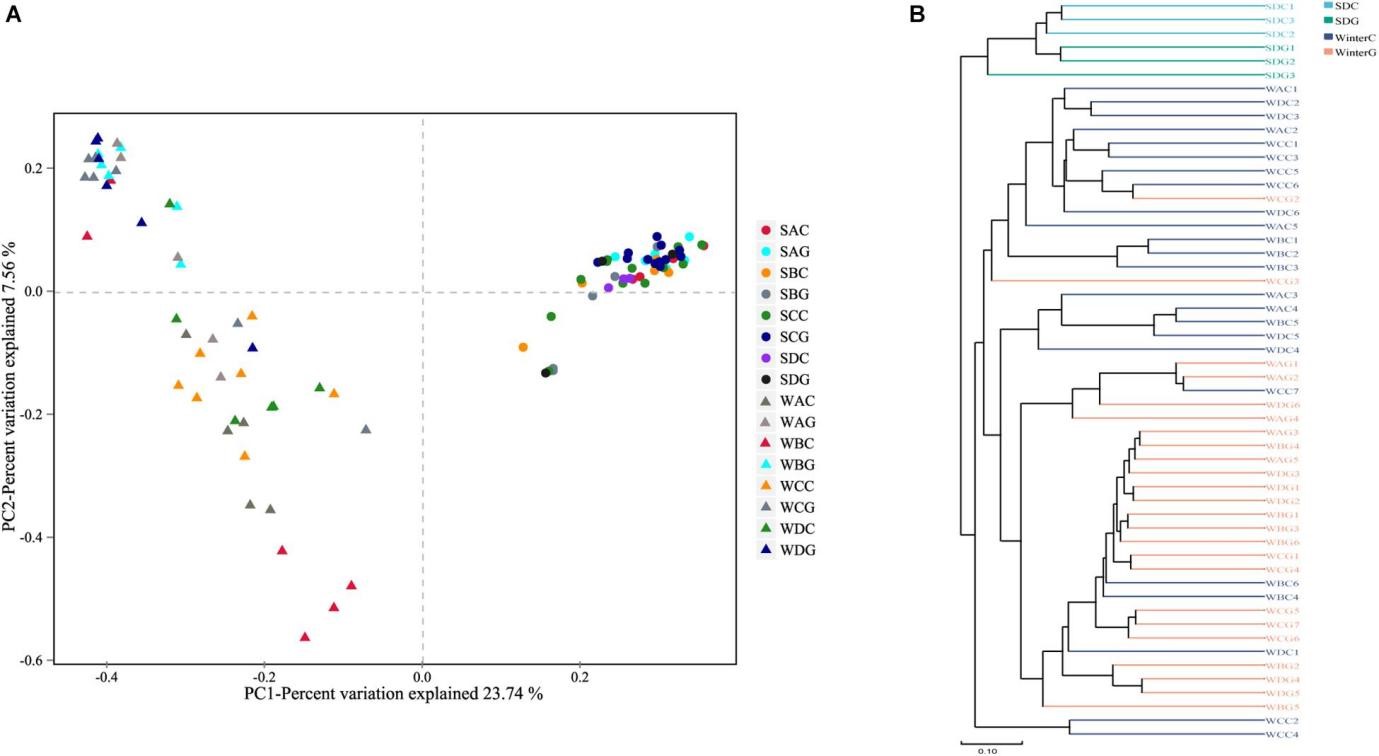
Hình 2. Biểu đồ hình hộp mô tả sự khác biệt về đa dạng beta giữa các mẫu đường ruột và gan tụy. (A) Tôm vào mùa hè; (B) Tôm vào mùa đông. Sự đa dạng beta của hệ sinh vật nấm đường ruột và gan tụy. (A) Phân tích tọa độ chính (PcoA) dựa trên khoảng cách Bray–Curtis. (B) Một chương trình dendro phân cụm được xây dựng bằng phương pháp nhóm cặp không trọng số với giá trị trung bình số học (UPGMA).
Theo Juan Li, Hai Jiang, Linmiao Li, Xiujian Zhang, Jinping Chen
Nguồn: https://www.frontiersin.org/articles/10.3389/fmicb.2019.00889/full
Biên dịch: Nguyễn Thị Quyên – Tôm Giống Gia Hóa Bình Minh
TÔM GIỐNG GIA HÓA – CHÌA KHÓA THÀNH CÔNG
Xem thêm:
- Lợi Ích Của Carotenoid Tự Nhiên Từ Paracoccus Carotinifaciens Đối Với Màu Sắc Và Hệ Miễn Dịch Của Tôm Thẻ Chân Trắng
- Tỉ Lệ Bao Nhiêu Mới Là Mức Tối Ưu Của Bột Cá Và Methionine Trong Chế Độ Ăn Của Tôm Giống?
- Phụ Gia Thức Ăn Phytogen: Tỏi Và Quế Có Thể Cải Thiện Sức Khỏe Và Hiệu Suất Tăng Trưởng Ở Tôm

 English
English