Tóm tắt
Amoniac là một tác nhân căng thẳng chính trong môi trường nuôi trồng thủy sản. Stress amoniac lâu dài có thể ảnh hưởng đến sự tăng trưởng bình thường, và cũng làm tăng nguy cơ mắc các bệnh khác nhau. Để tìm hiểu cơ chế mà amoniac gây ra sự bùng phát của bệnh tôm, các phương pháp tiếp cận phiên mã và chuyển hóa đã được sử dụng để phân tích các biểu hiện khác biệt của các gen trong tế bào máu và các chất chuyển hóa khác nhau trong huyết thanh của tôm thẻ chân trắng Litopenaeus vannamei khi phơi nhiễm với amoniac. Phân tích phiên mã cho thấy 17 gen liên quan đến quá trình chết rụng tế bào, 7 gen liên quan đến thực bào, 10 gen liên quan đến miễn dịch và 7 gen liên quan đến chu kỳ tế bào và chuyển hóa lipid cho thấy các biểu hiện khác biệt sau khi phơi nhiễm với amoniac. Phân tích chuyển hóa trên huyết thanh cho thấy 25 chất chuyển hóa phân biệt được xác định trong các mẫu ion dương và âm. Chúng tham gia vào quá trình chuyển hóa purine, chuyển hóa axit amin và lipid. Tiêm cho tôm bình thường hai chất chuyển hóa được điều chỉnh tăng triethanolamine và oxypurinol có thể gây ra quá trình chết rụng ở tôm bình thường. Phơi nhiễm với amoniac làm giảm đáng kể tổng số lượng tế bào máu và tăng đáng kể tỷ lệ tế bào apoptotic ở tôm. Những kết quả này cho thấy rằng việc phơi nhiễm với amoniac làm tăng quá trình chết rụng của tế bào máu, ảnh hưởng đến khả năng miễn dịch của tôm, và do đó gây ra sự nhạy cảm với nhiễm trùng gây bệnh. Những dữ liệu này sẽ giúp chúng ta hiểu được cơ chế của stress amoniac dẫn đến suy giảm miễn dịch ở tôm.
1. Giới thiệu
Tôm thẻ chân trắng Litopenaeus vannamei là một loài tôm quan trọng trong nuôi trồng thủy sản. Nó được nuôi rộng rãi nhất trên toàn thế giới do sinh trưởng nhanh, khả năng kháng bệnh, và khả năng thích ứng với môi trường nuôi mật độ cao (Cuzon và cộng sự, 2004; Kim và cộng sự, 2014). Tuy nhiên, với việc mở rộng quy mô canh tác và thúc đẩy chế độ nuôi mật độ siêu cao, môi trường nuôi trồng thủy sản bắt đầu xấu đi, và nhiều dịch bệnh xảy ra hơn, ảnh hưởng nghiêm trọng đến sản lượng và chất lượng của tôm thẻ chân trắng (Alagappan và cộng sự, 2010; Cuzon và cộng sự, 2004). Sự bùng phát dịch bệnh của tôm thường là kết quả của sự tương tác giữa các mầm bệnh, vật chủ và môi trường (Kautsky và cộng sự, 2000). Amoniac là yếu tố môi trường chính trong hệ thống nuôi cấy thủy sản, gây ra mối đe dọa lớn đối với sự sống của tôm (Cobo và cộng sự, 2014).
Trong môi trường nước, mưa và gió mang amoniac từ thức ăn dư thừa, phân của động vật, phân hủy chất rắn hữu cơ, amoniac dư thừa được tạo ra trong sản xuất nông nghiệp xung quanh vào bể nuôi trồng thủy sản (Kuhn và cộng sự, 2010). Amoniac thường có mặt ở các trạng thái ion hóa (NH4+) và không ion hóa (NH3) trong nước (Emerson và cộng sự, 1975; Fromm và Gillette, 1968), trong khi NH3 khuếch tán dễ dàng qua màng tế bào và vào hemolymph. Tỷ lệ NH4+ / NH3 có thể bị ảnh hưởng bởi pH, nhiệt độ và độ mặn (Chen và Chin, 1989; Kir và cộng sự, 2004; Lin và Chen, 2001). Các nghiên cứu cho thấy nồng độ amoniac cao có thể ức chế sự phát triển của tôm, tăng tần suất lột xác và dẫn đến tỷ lệ chết cao (Chen và Lin, 1992; Wickins, 1976). Ngoài ra, amoniac cũng có thể ảnh hưởng đến áp suất thẩm thấu, trạng thái sinh lý của mang và gan tụy (Lin và cộng sự, 1993; Lu và cộng sự, 2016). Nồng độ amoniac quá mức trong nước nuôi trồng thủy sản có thể ức chế hệ thống miễn dịch của tôm và tăng khả năng L. vannamei dễ bị nhiễm Vibrio hơn khi bị stress amoniac (Liu và Chen, 2004; Qiu và cộng sự, 2008). Do đó, việc hiểu một cách có hệ thống cơ chế phân tử của tôm thẻ L. vannamei đáp ứng với stress amoniac rất có giá trị. Trong nghiên cứu này, các chất chuyển hóa đã được sử dụng để nghiên cứu những thay đổi trong các chất chuyển hóa huyết thanh của tôm thẻ L. vannamei khi bị stress amoniac và bộ phiên mã được sử dụng để phân tích các đặc điểm biểu hiện gen của tế bào máu khi bị stress amoniac.
2. Vật liệu và phương pháp
2.1. Mẫu tôm
Tôm thẻ chân trắng L. vannamei khỏe mạnh nuôi trong bể cá của Viện Hải dương học, Viện Hàn lâm Khoa học Trung Quốc, Thanh Đảo, với trọng lượng trung bình 16,7g ± 5,8g, đã được sử dụng cho các thí nghiệm phơi nhiễm với amoniac. Trước khi thí nghiệm 3 ngày, tôm đã thích nghi ở 25 ± 1ºC với độ mặn 30‰ và pH 7,5 ± 0,1. Tất cả tôm được cho ăn 2 lần/ ngày vào lúc 8:00 sáng và 17:00 chiều với thức ăn viên thương mại (Công ty thức ăn chăn nuôi Dale, Yên Đài, Trung Quốc).
2.2. Phơi nhiễm với amoniac và thu thập mẫu
Dung dịch dự trữ amoniac-N (10 g/ L) được điều chế bằng cách hòa tan 38,19 g NH4Cl trong 1L nước cất (Liu và Chen, 2004). Nó được sử dụng để điều chỉnh nồng độ amoniac-N trong nước biển. Sau khi thích nghi, tôm được chia ngẫu nhiên thành 2 nhóm, nhóm phơi nhiễm với amoniac và nhóm đối chứng, mỗi nhóm có ba bể, 15 con trong mỗi bể với 50L nước biển. Nồng độ amoniac-N trong nhóm phơi nhiễm với amoniac được duy trì ở mức 20 mg/ L và nhóm đối chứng được nuôi trong cùng điều kiện mà không cần bổ sung amoniac. Trong quá trình thí nghiệm, nồng độ amoniac-N được đo và điều chỉnh sau mỗi 12 giờ bằng thuốc thử amoniac HACH (phương pháp axit salicylic, vận hành thủ công tham khảo) và dụng cụ quang kế cầm tay DR/ 850 (HACH, Hoa Kỳ). Nồng độ amoniac trong nước đã được điều chỉnh kịp thời để đảm bảo sự ổn định. Các điều kiện canh tác giống như mô tả ở trên. Sau 7 ngày thí nghiệm, 24 con đã được chọn ngẫu nhiên từ nhóm thử nghiệm và nhóm đối chứng tương ứng. Khoảng 400 mL hemolymph được thu thập từ mỗi con tôm tại xoang bụng nằm ở đoạn bụng đầu tiên với cùng một thể tích dung dịch thuốc chống đông máu (27 mM natri citrat, 336 mM NaCl, glucose 115 mM, 9 mM EDTA, pH 7) (Rodriguez và cộng sự, 1995). Hemolymph từ hai con được trộn thành một mẫu, và các tế bào máu và huyết thanh được tách ra bằng cách ly tâm ở 4ºC, 1000 g trong 5 phút. Toàn bộ 12 mẫu huyết thanh từ nhóm thí nghiệm hoặc nhóm đối chứng được bảo quản ở -80ºC để phân tích chất chuyển hóa. Các tế bào máu được bảo quản trong nitơ lỏng để phân tích phiên mã thêm. Ngoài ra, hemolymph của 5 con khác từ nhóm thí nghiệm và nhóm đối chứng đã được thu thập với cùng một phương pháp để đếm tổng số tế bào máu.
2.3. Phân tích bảng điểm
Các tế bào máu được thu thập từ 24 con trong mỗi nhóm được chỉ định ngẫu nhiên cho ba lần lặp lại. Các tế bào máu đã được thu thập để trích xuất RNA và phân tích giải trình tự phiên mã.
2.3.1. Phân lập RNA và giải trình tự illumina
Tổng RNA được chiết xuất từ các mẫu tế bào máu bằng thuốc thử RNAiso Plus (TaKaRa, Nhật Bản) theo hướng dẫn của nhà sản xuất. Chất lượng của tổng RNA được đánh giá bằng điện di trên gel agarose 1,0% và được định lượng bằng Máy đo quang phổ NanoDrop 2000 (Thermo Fisher Scientific Inc., Hoa Kỳ) để đảm bảo chất lượng cao của RNA. DNase I (Thermo scientific, Hoa Kỳ) đã được sử dụng để loại bỏ DNA bộ gen khỏi tổng số RNA.
Sau khi tổng số RNA được tách chiết, mRNA nhân chuẩn được làm giàu bằng các hạt Oligo (dT). Sau đó, mRNA đã làm giàu được phân mảnh thành các đoạn ngắn bằng cách sử dụng bộ đệm phân mảnh và phiên mã ngược thành cDNA với các mồi ngẫu nhiên. CDNA sợi thứ hai được tổng hợp thông qua việc thêm DNA polymerase I, RNase H, dNTP và bộ đệm. Sau đó, các mảnh cDNA được tinh chế bằng kit chiết xuất QiaQuick PCR, cuối cùng được sửa chữa, poly (A) được thêm vào và được gắn vào bộ điều hợp giải trình tự Illumina. Các sản phẩm liên kết có kích thước được lựa chọn bởi điện di gel agarose, khuếch đại PCR và giải trình tự bằng Illumina HiSeqTM 2000 bởi công ty công nghệ sinh học Gene Denovo (GENE DENOVO, Quảng Châu).
2.3.2. Phân tích tin sinh học
Trình tự bộ điều hợp, số lần đọc chất lượng thấp, RNA ribosome (rRNA) và các lần đọc trống đã bị xóa khỏi các lần đọc thô, và được chuyển thành các lần đọc sạch. Các lần đọc sạch chất lượng cao đã được ánh xạ tới bảng phiên mã tham chiếu bằng cách sử dụng công cụ căn chỉnh đọc ngắn Bowtie 2 (Li và cộng sự, 2009) và được chuyển đổi thành mảnh trên mỗi kilobase bảng phiên mã trên một triệu cặp cơ sở được sắp xếp (FPKM) (Mortazavi và cộng sự, 2008). Phân tích tương quan của 3 thí nghiệm song song cung cấp đánh giá độ tin cậy của kết quả thí nghiệm cũng như sự ổn định hoạt động. Phân tích thành phần chính (PCA) được thực hiện với các mô hình gói R (http://www.r-project.org/ ). Gói edgeR (http://www.rproject.org/ ) được sử dụng để xác định các gen biểu hiện khác nhau (DIG) trên 2 nhóm. Những gen có thay đổi 2 nếp gấp và tỷ lệ phát hiện sai (FDR) < 0,05 trong một so sánh được xác định là DEG quan trọng. Các DEG sau đó đã được phân tích làm giàu các chức năng của Bản thể học gen (GO) và các con đường dẫn tín hiệu của bộ bách khoa toàn thư về gen và bộ gen Kyoto (KEGG).
2.3.3. Phân tích định lượng real-time PCR của các gen được chọn từ bộ phiên mã
Để xác minh dữ liệu phiên mã, 6 DEG đã được chọn ngẫu nhiên để phân tích PCR định lượng thời gian thực. Gen housekeeping 18S rRNA được sử dụng làm tiêu chuẩn nội bộ (Sun và cộng sự, 2014). Thông tin về các mồi được sử dụng cho PCR thời gian thực đã được hiển thị trong Bảng 1. RNA được chiết xuất được sử dụng để tổng hợp cDNA bằng kit tổng hợp cDNA I PrimeScript™ với 6 mã phân tử ngẫu nhiên (TaKaRa, Nhật Bản). Phản ứng qPCR được thực hiện trong tổng thể tích 10 mL, chứa 5 mL SuperReal PreMix Plus (SYBR Green) (Tiangen, Trung Quốc), mồi trước 0,3 mM, mồi ngược 0,3 mM, mẫu cDNA 1 mL và nước không có RNase 3,4 mL. Chương trình được thiết lập như sau: 95ºC trong 15 phút; 40 chu kỳ 95ºC trong 15 giây, nhiệt độ ủ (thể hiện trong Bảng 1) trong 15 giây và 72ºC trong 20 giây, sử dụng Eppendorf Mastercycler® ep realplex (Eppendorf, Đức). Mức độ biểu hiện tương đối của gen đích được tính bằng phương pháp 2–rrCT (Livak và Schmittgen, 2001).
Bảng 1. Thông tin về mồi được sử dụng cho PCR thời gian thực.
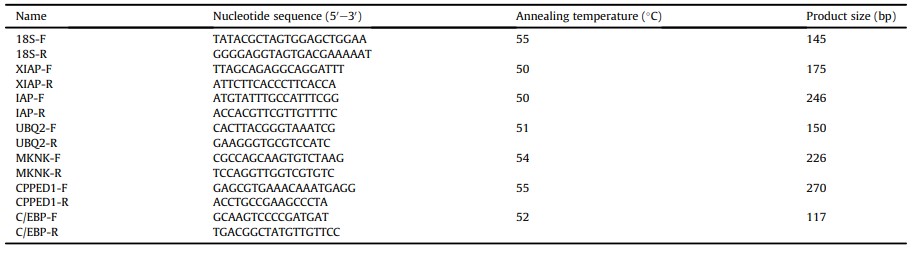
2.4. Phân tích chuyển hóa
Huyết thanh của tôm từ nhóm thí nghiệm và đối chứng đã được sử dụng để phân tích chất chuyển hóa. Mỗi nhóm có 12 mẫu lặp lại. Các mẫu đông lạnh được rã đông ở 4ºC và 100 mL của mỗi mẫu được đồng nhất với 400 mL dung môi lạnh (methanol/ acetonitril 1/4 1:1, v/v), và đặt ở 20ºC trong 30 phút, sau đó ly tâm ở 14.000 g ở 4ºC trong 20 phút. Chất nổi trên bề mặt được chuyển đến một ống ly tâm mới, và sau đó được đông khô trong một bộ cô đặc chân không. Các mẫu khô được hòa tan và đồng nhất trong dung dịch nước acetonitril (acetonitril: nước 1/4 1: 1, v/ v) và ly tâm ở 14.000g ở 4ºC trong 15 phút. Chất nổi trên bề mặt được sử dụng để phân tích quang phổ khối lượng. Phân tích trao đổi chất được thực hiện tại công ty TNHH công nghệ protein thực nghiệm Thượng Hải (APTBIO, Trung Quốc).
2.4.1. Điều kiện sắc ký lỏng
Sắc ký lỏng hiệu suất cực cao (UHPLC) (Agilent, Hoa Kỳ) và cột sắc ký HILIC (ACQUITY UPLC BEH Amide 1,7 mm, cột 2,1 mm 100 mm, Waters, Hoa Kỳ) đã được sử dụng để phân tích sắc ký. Điều kiện làm việc cho cột được đặt như sau: nhiệt độ ở 25ºC, tốc độ dòng chảy ở 0,3 mL/ phút, thể tích phun ở 2 mL. Một pha di động (A) có chứa amoni axetat (25 mM) và amoniac nước (25 mM) trong nước; Pha di động B (B) là acetonitril. Chương trình giải nén gradient kéo dài 20 phút như sau: Sau khi bắt đầu trước với 85% di động giai đoạn B trong 1 phút, thay đổi tuyến tính giai đoạn B di động thành 65% từ 1 đến 12 phút, và sau đó lên 40% từ 12 đến 12,1 phút, ở mức 40% trong 3 phút và trở lại 85% từ 15 đến 15,1 phút, tiếp theo là giữ ở mức 85% cho đến 20 phút. Các mẫu được đặt trong bộ lấy mẫu tự động 4ºC trong toàn bộ quá trình. Cứ 5 mẫu kiểm tra chất lượng lại được thiết lập để đánh giá độ ổn định của hệ thống.
2.4.2. Điều kiện khối phổ kế
Sau khi các mẫu được tách ra bằng UHPLC, phép đo phổ khối lượng được thực hiện với máy quang phổ khối lượng Triple TOF 6600 (AB SCIEX, HOA KỲ). Phép đo phổ khối lượng ion hóa phun điện tử (ESI) (MS) được sử dụng để phát hiện ở cả chế độ ion dương và âm. Các điều kiện nguồn ESI như sau: Ion Source Gas1 (Gas1): 60 psi, Ion Source Gas2 (Gas2): 60 psi, Curtain gas (CUR): 30 psi, nhiệt độ nguồn: 600ºC, IonSapary Voltage Floating (ISVF) ± 5500V (Chế độ dương và âm); TOF MS quét m/ z phạm vi: 60-1000 Da, quét ion sản phẩm m/ z phạm vi: 25-1000 Da, thời gian tích lũy quét TOF MS 0,20 s/ quang phổ, thời gian tích lũy quét ion sản phẩm 0,05s/ quang phổ; Phổ khối lượng thứ cấp thu được bằng cách sử dụng thu nhận phụ thuộc thông tin (IDA) và với chế độ nhạy cao, Tiềm năng khử nhiễu (DP): ±60V (Chế độ dương và âm), Năng lượng va chạm: 35 ± 15 eV, cài đặt IDA như sau: Loại trừ các đồng vị trong 4 Da: 6 ion ứng cử viên để theo dõi mỗi chu kỳ.
2.4.3. Phân tích thống kê
Dữ liệu gốc đã được ProteoWizard chuyển đổi sang định dạng “.mzML”. Phần mềm XCMS được sử dụng để khớp đỉnh, căn chỉnh thời gian duy trì và khu vực đỉnh. Nhận dạng chất chuyển hóa được thực hiện bằng cách tìm kiếm cơ sở dữ liệu của công ty (APTBIO, Trung Quốc) trong kết quả trùng khớp khối lượng chính xác (50% trong nhóm đã bị xóa khỏi dữ liệu thu được từ XCMS. Phần mềm ứng dụng SIMCA-P 14.1 (Umetrics, Umea, Thụy Điển) được sử dụng để nhận dạng mẫu, dữ liệu được xử lý trước bằng chia tỷ lệ pareto và phân tích thống kê đa chiều, và phân tích thành phần chính không giám sát (PCA) đã được thực hiện.
2.4.4. Phân tích tin sinh học
Để đánh giá tính hợp lý của các chất chuyển hóa ứng cử viên, mối quan hệ giữa các mẫu và các mẫu biểu hiện của các chất chuyển hóa trong các mẫu khác nhau đã được hiển thị toàn diện và trực quan hơn. Sự khác biệt đáng kể về chất của biểu thức chất chuyển hóa đã được sử dụng để phân tích cụm phân cấp trên từng nhóm mẫu. Các chất chuyển hóa vi phân đã được gửi đến cơ sở dữ liệu KEGG (http://www.kegg.jp/kegg/pathway.html ) để phân tích con đường làm giàu.
2.5. Tổng số tế bào máu ở tôm
Hemolymph của năm con từ nhóm thí nghiệm và nhóm đối chứng được thu thập bằng phương pháp tương tự như được mô tả trước đây trong phần 2.2. Tổng số tế bào máu được tái sử dụng và pha loãng 10 lần trong hỗn hợp thể tích bằng nhau của môi trường L15 và thuốc chống đông máu. Tổng số lượng tế bào máu tính bằng đơn vị thể tích (mL) được phân tích bằng BD FACSAria II Flow Cytometer (Bection Dickinson, Hoa Kỳ). Đối với mỗi mẫu, 10.000 tế bào đã được tính. Nồng độ tế bào máu được tính theo tốc độ dòng chảy và thời gian ghi lại.
2.6. Xét nghiệm apoptosis cho các tế bào máu
Các tế bào máu apoptotic đã được nghiên cứu bằng bộ dụng cụ phát hiện quá trình apoptosis Annexin V-FITC/ PI (công nghệ sinh học Jiancheng Nam Kinh, Trung Quốc). 10 con từ mỗi nhóm được lấy mẫu ngẫu nhiên lần lượt vào 0, 1, 3, 5 và 7 ngày sau khi phơi nhiễm với amoniac. Hemolymph được thu thập từ xoang bụng nằm ở đoạn bụng đầu tiên với thể tích bằng nhau của dung dịch Alsever biến đổi thuốc chống đông máu. Tế bào máu được phân lập bằng cách ly tâm ở 4ºC, 1000 g trong 10 phút. Tế bào máu dạng viên được rửa bằng dung dịch chống đông máu 2 lần và được tái sử dụng ở khoảng 1 106 tế bào/ mL trong 500 mL đệm liên kết, và sau đó 5 mL Annexin V-FITC và 5 mL dung dịch làm việc PI đã được thêm vào. Huyền phù tế bào được trộn và ủ trong 10 phút ở nhiệt độ phòng trong bóng tối và ngay lập tức được phân tích bởi hệ thống tế bào học dòng chảy BD FACSAria II (Bection Dickinson, Hoa Kỳ). Đối với mỗi mẫu, 10.000 tế bào đã được tính. Các tế bào nhuộm màu âm tính với cả hai đầu dò đại diện cho các tế bào sống. Các tế bào nhuộm màu dương tính với Annexin V-FITC và âm tính với PI là các tế bào apoptotic sớm. Các tế bào nhuộm màu dương tính với cả Annexin V-FITC và PI đang ở giai đoạn cuối của quá trình apoptosis, trải qua hoại tử hoặc đã chết. Tỷ lệ tế bào apoptotic (AR) được định nghĩa là tỷ lệ phần trăm của tế bào máu apoptotic (Guo và cộng sự, 2013).
2.7. Tác dụng của các chất chuyển hóa phân biệt đối với quá trình apoptosis của tế bào máu
Bốn chất chuyển hóa cho thấy sự điều hòa đáng kể trong huyết thanh của tôm khi phơi nhiễm với amoniac, bao gồm L-proline (Sigma, Hoa Kỳ), 1, 2-Di- (9Z-octadecenoyl)-sn-glycero-3- phosphocholine (Sigma, Hoa Kỳ), triethanolamine (Thượng Hải Hushi, Trung Quốc), oxypurinol (Sigma, Hoa Kỳ) đã được chọn để phân tích chức năng. Tôm có trọng lượng trung bình 13,9 ± 2,1g đã được sử dụng để xử lý chất chuyển hóa. Mỗi nghiệm thức trao đổi chất chứa 10 con và bốn nghiệm thức trao đổi chất đã được thiết lập. Mỗi con tôm được tiêm một chất chuyển hóa hòa tan trong PBS với liều 100 mg/ g tôm. Tôm trong nhóm đối chứng được tiêm cùng một lượng PBS vô trùng. Tất cả tôm được cho ăn và nuôi trong nước biển để quản lý định kỳ. Sau 4 giờ tiêm, tế bào máu của năm cá thể được thu thập từ mỗi nghiệm thức trao đổi chất và nhóm đối chứng xét nghiệm apoptosis với cùng một phương pháp như được mô tả trong phần 2.7.
2.8. Phát hiện vi khuẩn đang bám trong hemolymph của tôm và nước biển ao nuôi
Hemolymph của năm con được thu thập từ nhóm thí nghiệm và nhóm đối chứng theo quy trình được mô tả trong phần 2.2 và được sử dụng để phát hiện vi khuẩn in vivo. Mỗi mẫu được pha loãng trong PBS vô trùng với tỷ lệ 1: 9 (v/v). Pha loãng thập phân nối tiếp đã được chuẩn bị từ 10-1 đến 10-4 . Tổng cộng 100 mL mỗi lần pha loãng được gieo vào muối mật thiosulfate citrate môi trường thạch sucrose (TCBS) và môi trường thạch đậu nành tryptic (với 2,0% natri clorua, TSA), và sau đó chúng được ủ ở 30ºC trong 18 giờ. Đồng thời, các mẫu 1 mL nước biển của mỗi ao được thu thập từ các ao chứa nhóm thí nghiệm và nhóm đối chứng, và được xử lý như mô tả ở trên để đếm vi khuẩn. Các khuẩn lạc trên tấm TCBS và TSA được tính riêng để phân tích thống kê.
3. Kết quả
3.1. Phân tích phiên mã trên tế bào máu của tôm khi phơi nhiễm với amoniac
3.1.1. Đọc chú thích lắp ráp và trình tự
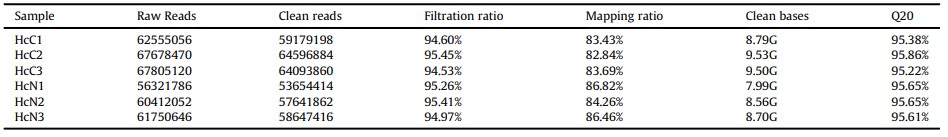
Tổng quan về thông tin trình tự của bảng phiên mã đã được liệt kê trong Bảng 2. Tổng cộng 53,654,414 đến 64,596,884 lần đọc sạch đã thu được cho mỗi mẫu, 82.84%-86.82% được so sánh so với bộ gen tham chiếu. Tất cả các lần đọc sạch được lắp ráp thành 61.283 unigenes, tỷ lệ GC là 40,92%, N50 là 1308 bp và chiều dài trung bình là 766 bp. Tất cả các unigenes đã được tìm kiếm dựa trên bốn cơ sở dữ liệu BLASTX. Tổng cộng 12,426 unigenes đã được nối với các protein đã biết trong cơ sở dữ liệu NR, 9,715 unigenes được nối với các tương đồng giả định trong cơ sở dữ liệu Swiss-Proton, 8,850 unigenes được chú thích trong cơ sở dữ liệu KOG và 6,935 unigenes được chú thích trong cơ sở dữ liệu KRA.
Bảng 2. Tổng quan về trình tự và lắp ráp bộ phiên mã tôm từ tôm thẻ chân trắng
3.1.2. Các gen biểu hiện khác biệt trong tế bào máu khi phơi nhiễm với amoniac
Tổng cộng có 516 gen biểu hiện khác biệt (DEGs) cho thấy các biểu hiện khác biệt trong tế bào máu của tôm khi phơi nhiễm với amoniac, bao gồm 145 DEG được điều chỉnh tăng và 371 DEG được điều chỉnh giảm (Bảng S1). Thông qua chú thích NR cho DEGs, chúng tôi nhận thấy rằng nhiều người trong số họ có liên quan đến quá trình apoptosis, thực bào, miễn dịch và chu kỳ tế bào và gen chuyển hóa lipid (Bảng 3). Trong số đó, 17 DEG liên quan đến quá trình chết rụng tế bào cho thấy các biểu hiện khác biệt sau khi phơi nhiễm với amoniac, bao gồm 12 gen liên quan đến ức chế quá trình chết rụng và 11 trong số đó có quy định thấp, 5 gen liên quan đến hoạt hóa quá trình chết rụng và ba trong số được điều chỉnh tăng, điều này chỉ ra rằng quá trình chết rụng tế bào máu có thể được kích hoạt ở tôm sau khi phơi nhiễm với amoniac. 7 DEG liên quan đến thực bào cho thấy sự điều hòa đáng kể trong tế bào máu của tôm khi phơi nhiễm với amoniac. 6 trong số 10 DEG liên quan đến khả năng miễn dịch cho thấy biểu hiện được điều chỉnh theo quy định và 4 DEG còn lại cho thấy biểu hiện giảm quy định trong nhóm phơi nhiễm với amoniac. 3 gen được điều chỉnh xuống đã tham gia vào con đường PI3K. 7 DEG liên quan đến chu kỳ tế bào và chuyển hóa lipid, chẳng hạn như tổng hợp axit béo, tổng hợp phospholipid và các quá trình khác liên quan đến tổng hợp thành phần tế bào, tất cả đều được kiểm soát trong nhóm phơi nhiễm với amoniac
Bảng 3. Các DEG chính liên quan đến quá trình apoptosis, miễn dịch, thực bào và chu kỳ tế bào và chuyển hóa lipid.



3.1.3. Xác minh dữ liệu bảng phiên mã bằng thử nghiệm qPCR
Sáu gen bao gồm 3 gen điều hòa xuống (XIAP, chất ức chế liên kết X của protein apoptosis; IAP, chất ức chế protein apoptosis; UBQ2, polyubiquitin 2) và 3 gen được điều chỉnh lên (MKNK, lập bản đồ serine/ threonine tương tác kinase; CPPED1, giống serine/ threonineprotein phosphatase CPPED1; C/ EBP, protein liên kết CCAAT/ chất tăng cường) đã được chọn để xác nhận các mẫu biểu hiện bằng thử nghiệm qPCR. Kết quả cho thấy hồ sơ biểu hiện của các gen này cho thấy xu hướng tương tự với các gen trong phân tích bảng phiên mã (Hình 1).
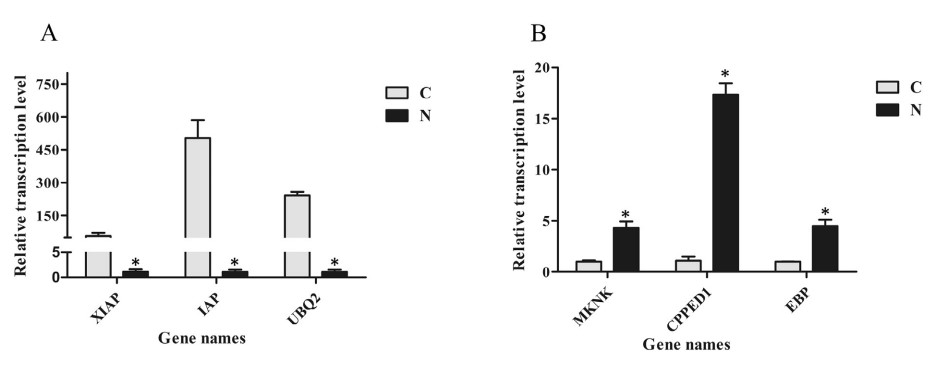
Hình 1. Định lượng kết quả phân tích PCR thời gian thực của 6 gen. XIAP, E3 ubiquitin-protein ligase XIAP; IAP, chất ức chế protein apoptosis; UBQ2, polyubiquitin 2; MKNK, bản đồ kinaseinteracting serine/threonine; CPPED1, serine/ threonine-protein phosphatase CPPED1 giống như; C/EBP, CCAAT/protein liên kết với chất tăng cường. C, nhóm đối chứng; N, nhóm tiếp xúc với amoniac. Các thanh dọc đại diện cho giá trị trung bình ± S.E. (n 1/4 3). Các ngôi sao (*) cho thấy sự khác biệt đáng kể về mức độ biểu hiện gen giữa nhóm căng thẳng amoniac và nhóm đối chứng ở P < 0,05.
Theo Fei Liu, Shihao Li, Yang Yu, Mingzhe Sun, Jianhai Xiang, Fuhua Li
Biên dịch: Nguyễn Thị Quyên – Tôm Giống Gia Hóa Bình Minh
Nguồn: https://www.sciencedirect.com/science/article/abs/pii/S0045653519319964
TÔM GIỐNG GIA HÓA – CHÌA KHÓA THÀNH CÔNG
Xem thêm:
- Ủ Chua Phụ Phẩm Sau Chế Biến Của Cá Rô Phi (TPWS): Một Nguyên Liệu Thay Thế Cho Khẩu Phần Ăn Của Tôm Thẻ Chân Trắng Litopenaeus vannamei (Boone, 1931) Trong Các Hệ Thống Biofloc Và Nước Sạch
- Rà Soát Lại Bệnh Vibrio Trong Nuôi Tôm
- Ảnh Hưởng Của Probiotics Đến Sự Phân Hủy Amoniac Trong Nghiên Cứu In Vitro Và Trong Ao Nuôi Tôm Thẻ Chân Trắng

 English
English 中文 (中国)
中文 (中国)